|
Оценка коллатеральной активности CRISPR/Cas13a-рибонуклеазы на биоанализаторе Agilent 2100 Научно-исследовательский институт биомедицинской химии им. В. Н. Ореховича, Москва, 119121, ул. Погодинская 10; *e-mail: kurbatovl@mail.ru Ключевые слова: CRISPR/Cas13-рибонуклеаза; коллатеральная активность; детекция; Bioanalyzer 2100 DOI: 10.18097/BMCRM00169 ВВЕДЕНИЕ
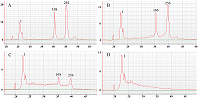
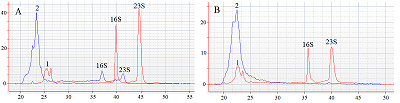
CRISPR/Cas13a-pибонуклеаза является центральным компонентом ультрачувствительной системы детекции РНК, известной как SHERLOCK (Specific High Sensitivity Enzymatic Reporter unLOCKing) [1-3]. Данная система основана на способности рибонуклеазы селективно распознавать таргетную молекулу РНК после формирования дуплекса между спейсером направляющей РНК (нРНК) и комплементарным ему участком РНК-мишени. После связывания и расщепления таргетной молекулы рибонуклеаза теряет свою специфичность и приобретает способность деградировать любые молекулы РНК, то есть приобретает так называемую «коллатеральную активность». Детекция коллатеральной активности, как правило, осуществляется по деградации добавленных в реакционную смесь коротких молекул РНК, меченых флуорофором и тушителем (РНК-субстрат или РНК-репортеры), посредством регистрации увеличения флуоресцентного сигнала [1-3]. Также при замене флуорофора на биотиновую метку анализ может быть проведен в формате тест-полосок, что открывает перспективы для полевой диагностики бактериальных и вирусных патогенов [4]. В сочетании с изотермической амплификацией целевого фрагмента РНК чувствительность биосенсоров на основе системы SHERLOCK находится на уровне единичных молекул РНК [5]. В лабораторных условиях детекцию коллатеральной активности Cas13a-нуклеазы проводят путём регистрации флуоресценции с использованием флуоресцентно-меченых РНК-репортеров [1-5]. Это требует наличия в лаборатории соответствующего оборудования для измерения интенсивности флуоресценции. В то же время в лабораториях, работающих с нуклеиновыми кислотами, получило широкое распространение использование биоанализаторов Agilent 2100 («Agilent», США), которые рутинно применяются для анализа качества препаратов суммарной РНК. Биоанализатор Agilent 2100 представляет собой автоматизированное устройство для проведения капиллярного электрофореза на чипах, выдающее результаты в виде электрофореграмм в координатах «флуоресценция (детекция нуклеиновых кислот проводится с использованием флуоресцентных красителей со сродством к ДНК или РНК) – время достижения аналитом позиции детектора». При анализе суммарной РНК доминантой на электрофореграммах являются пики рибосомной РНК (рРНК). Неспецифическое расщепление РНК в результате приобретённой Cas13a коллатеральной активности приводит, в том числе, и к деградации рРНК, что должно проявляться в визуально наблюдаемом уменьшении её пиков на электрофореграмме. Целью работы было показать возможность использования биоанализаторов Agilent 2100 для качественной оценки коллатеральной активности рекомбинантной CRISPR/Cas13a-рибонуклеазы и подобрать оптимальные условия для такого тестирования, используя нРНК, узнающую последовательность в 16S рРНК E. coli. Детекция коллатеральной активности Cas13a с использованием биоанализатора Agilent 2100 представляется оправданной в случаях, когда не требуется высокая чувствительность и специфичность метода – например, при скринировании образцов рибонуклеазы, полученных в различных условиях, из различных источников или разными способами очистки. МАТЕРИАЛЫ И МЕТОДЫ Оригинальная методика получения рекомбинантной нуклеазы Cas13a одностадийной очисткой методом металл-хелатной хроматографии после гетерологической экспрессии подробно описана в работе [6]. Там же описаны процедуры выделения суммарной РНК из клеток E. coli штамма XL1 Blue, синтеза нРНК, специфичной к участку 16S рРНК E. coli, а также методы определения их концентраций и концентрации белка. В рамках данной работы наличие коллатеральной активности препаратов Cas13a предварительно тестировали с помощью набора RNaseAlert® Lab Test Kit v2 («Thermo Fisher Scientific», США), использующего флуоресцентно меченые РНК-репортёры. Все остальные реактивы были приобретены в «Sigma-Aldrich» (США) и соответствовали качеству «ACS grade» или выше. Для приготовления растворов использовалась вода, свободная от нуклеаз («Sigma-Aldrich»). Оценку влияния рибонуклеазной активности на профиль суммарной РНК на электрофореграммах осуществляли добавлением РНКазы А из набора RNaseAlert® Lab Test Kit v2 к раствору тРНК в буфере 1X RNaseAlert Lab Test Buffer (далее буфер А). Аликвоту стокового раствора РНКазы А с активностью 0.03 Ед/мл предварительно разводили тысячекратно буфером А и добавляли 1 мкл раствора к пробе суммарной РНК объемом 50 мкл. Смесь инкубировали 10 мин, 60 мин или 120 мин при 37°С. Конечная концентрация суммарной РНК в пробе составляла 100 нг/мкл, активность РНКазы А – 6∙10-9 Ед/мкл. Перед электрофоретическим анализом пробы разводили 100-кратно водой, свободной от нуклеаз. Для детекции коллатеральной активности препарат рекомбинантной Cas13a-нуклеазы после очистки разбавляли реакционным буфером, содержащим 40 мМ Трис-HCl (pH 7,3), 60 мМ NaCl и 6 мМ MgCl2 (далее буфер R1) до требуемой концентрации, после чего 1 мкл полученного раствора белка смешивали с 1 мкл водного раствора нРНК и инкубировали 15 мин при 37°С для формирования комплекса нРНК/Cas13a. После инкубации пробы охлаждали на льду, центрифугировали 30 с при 3000 g и добавляли 1 мкл раствора суммарной РНК в воде. Объём пробы доводили до 4 мкл воды, свободной от нуклеаз, при этом конечная концентрация нРНК равнялась 450 нМ, тРНК – 5 нг/мкл, конечная концентрация Cas13 варьировала от 45 нМ до 180 нМ, а конечная композиция реакционного буфера была соответственно 10 мМ Трис-HCl (pH 7,3), 15 мМ NaCl и 1,5 мМ MgCl2 (далее буфер R2). Пробу инкубировали в течение 1 ч при 37°С, после чего снова центрифугировали 30 с при 3000 g. Контрольные пробы готовили и обрабатывали так же, за исключением того, что вместо раствора Cas13-нуклеазы и/или нРНК добавляли соответствующий объем воды, свободной от нуклеаз. В специально оговорённых случаях пробы могли содержать ингибитор РНКаз Murine RNase Inhibitor («New England Biolabs», Великобритания) в концентрации 1.6 Ед/мкл. Анализ проб проводили с помощью прибора Agilent 2100, используя чипы из набора Agilent RNA 6000 Pico Kit. Подготовку реактивов, чипов и прибора проводили в соответствии с протоколом фирмы-производителя. В каждую ячейку чипа вносили 1 мкл пробы. МАТЕРИАЛЫ И МЕТОДЫ На рисунке 1 представлен результат инкубирования суммарной РНК с РНКазой А. Электрофоретический профиль исходной тРНК характеризуется наличием двух пиков, соответствующих 16S и 23S рРНК, которые проходят позицию детектора биоанализатора приблизительно на 40 с и 45 с электрофореза (рис. 1А). Пик, появляющийся на ≈26 секунде, представляет флуоресцентный лидирующий маркер, входящий в набор Agilent RNA 6000 Pico Kit и добавляемый как контроль в анализируемые пробы в соответствии с рекомендацией производителя. Как и ожидалось, деградация суммарной РНК рибонуклеазой А приводит к заметному снижению высоты пиков 16S и 23S рРНК уже через 10 мин инкубации (рис. 1B), а через 1 ч их высота снижается драматически (рис. 1C). Пики рРНК полностью исчезают при двухчасовой инкубации (рис. 1D). Таким образом, присутствие неспецифической рибонуклеазной активности в анализируемой пробе может быть однозначно детектировано на биоанализаторе Agilent 2100 по уменьшению высоты пиков рРНК во временном интервале инкубации до 1 ч. При этом стоит также отметить, что внесение в пробу РНКазы А сдвигает положение пиков рРНК (приблизительно на 5 с) и маркера (на ≈2 с) на оси времени (рис. 1 А,B). Это указывает на то, что усложнение композиции стартовой зоны из-за внесения РНКазы А влияет на скорость электрофоретической миграции компонентов пробы, увеличивая их приблизительно на 10%.
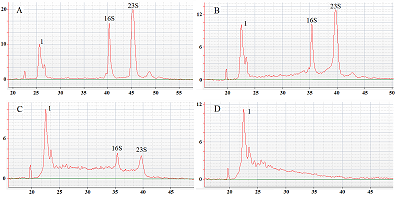
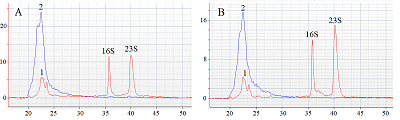
Характерные электрофореграммы для тРНК, нРНК, а также их смесей, в том числе с Cas13a, после инкубации в буфере R2 в течении 1 ч представлены на рис. 2 (А-D). Как можно видеть, и в этом случае электрофоретический профиль суммарной РНК характеризуется наличием двух пиков, появляющихся на приблизительно 40 с и 45 с электрофореза и соответствующих 16S и 23S рРНК (рис. 2 А). Присутствие в пробе нРНК в концентрации 450 нМ не оказывает существенного влияния на электрофоретическое поведение тРНК: положение и высота пиков 16S и 23S рРНК практически не меняются (рис. 2 С). Cas13a в концентрации 45 нМ (рис. 2 D) также не оказывала заметного влияния на электрофоретический профиль суммарной РНК, однако присутствие в пробе комплексов нРНК/Cas13a (концентрация Cas13a 45 нМ) приводило к изменению скорости электрофоретической миграции 16S и 23S рРНК – пики видны на ≈36 с и ≈39 с, при этом их высота уменьшается примерно на треть (в среднем, с приблизительно 70 условных единиц (у.е.) флуоресценции до приблизительно 45 у.е.), что может указывать на начало деградации рРНК. При этом высота пика нРНК практически не менялась.
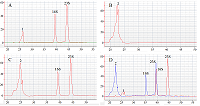
Увеличение концентрации Cas13a в пробе до 90 нМ не оказывало дальнейшего влияния на скорость электрофоретической миграции рРНК, но снижало высоту её пиков приблизительно на 40% (до 35-40 у.е. флуоресценции, рис. 2 С, 3 А). При этом наличие комплексов нРНК/Cas13a, формирующихся при этих концентрациях нРНК и Cas13a, вызывало значительное (14-кратное, до ≈5 у.е.) уменьшение высоты пиков рРНК и незначительное (около 30%) – высоты пика нРНК (рис. 3А). Скорость электрофоретической миграции рРНК при такой композиции компонентов пробы увеличивалась ещё больше. Снижение высоты пика нРНК (рис. 3А) может быть связано с увеличением доли нРНК в комплексе с Cas13a. Комплексы нРНК/Cas13a, скорее всего, имеют отличающуюся скорость электрофоретической миграции и не достигают детектора за типичное время электрофореза (55-65 с), в то время как многократное уменьшение высоты пиков рРНК указывает на её эффективную деградацию. Дальнейшие увеличение концентрации Cas13a до 180 нМ в присутствии нРНК приводило к полному исчезновению пиков рРНК (рис. 3 B); однако при этом Cas13a в этой концентрации в отсутствии комплексов нРНК/Cas13a также вызывала заметное (5-6-кратное, до 12 у.е. флуоресценции) уменьшение высоты пиков рРНК. Пик нРНК также уменьшался – его высота соответствовала в этом случае ≈25 у.е. флуоресценции (рис. 3Б). Это хорошо согласуется с возрастанием относительной доли нРНК в комплексе с Cas13 при увеличении концентрации последней и повышении эффективности деградации рРНК при возрастании количества комплексов нРНК/Cas13. Следует также отметить, что при данной концентрации Cas13 (180 нМ) скорость электрофоретической миграции рРНК ещё более возрастает даже в отсутствии нРНК (рис. 3Б).
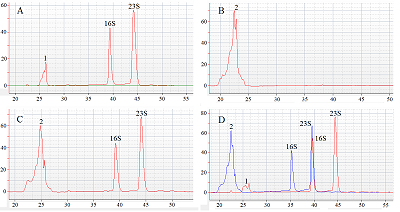
Одной из причин прогрессивного снижения высоты пиков рРНК при возрастании концентрации Cas13-нуклеазы, но в отсутствии комплексов нРНК/Cas13 и соответственно коллатеральной активности Cas13-нуклеазы, может быть загрязнение препарата Cas13 РНКазами E. coli. Для проверки этого предположения, мы добавили в реакционную смесь ингибитор РНКаз (Murine RNase Inhibitor) в концентрации, которая рекомендуется для подавления фоновой рибонуклеазной активности [1, 2]. Однако добавление ингибитора не оказало существенного влияния на высоту пиков рРНК (приблизительно 12 и 15 у.е. флуоресценции в отсутствии и присутствии ингибитора соответственно; рис. 4).
ОБСУЖДЕНИЕ В предлагаемом подходе к детекции коллатеральной активности Cas13a используется электрофоретический анализ суммарной РНК, которая совмещает в себе как мишень (участок 16S рРНК), так и РНК-субстрат, который может быть использован для детектирования коллатеральной активности Cas13a-нуклеазы по изменению высоты пиков 16S и 23S рРНК. Как показывают результаты с РНКазой А, присутствие рибонуклеазной активности приводит к прогрессивному уменьшению высоты пиков рРНК. В случае Cas13a, такая активность может включать как специфическую, связанную с расщеплением 16S рРНК рядом с последовательностью-мишенью, так и неспецифическую (коллатеральную), приводящую к деградации молекул РНК независимо от их последовательности. Поскольку 23S рРНК не имеет в своём составе последовательности-мишени, уменьшение высоты её пика на электрофореграмме однозначно указывает на появление у Cas13a коллатеральной активности. При этом в наблюдаемую деградацию 16S рРНК может вносить вклад и специфическая активность Cas13a. Применение в нашей работе буфера R2 вместо R1, который используется при детекции деградации Cas13a-нуклеазой флуоресцентно-меченных РНК-репортеров после обретения ею коллатеральной рибонуклеазной активности [1, 2], было связано с тем, что при приготовлении проб в буфере R1 высота пиков рРНК на электрофореграммах существенно снижалась даже в контрольных образцах, что затрудняло оценку коллатеральной активности. Скорее всего это связано с тем, что чипы Agilent RNA 6000 Pico оптимизированы производителем для анализа РНК, находящейся в растворе с низкой ионной силой. Действительно, последовательные 2-кратные разведения буфера R1 водой показали, что при его 4-кратном разведении (буфер R2) на электрофореграммах появляются хорошо выраженные пики рРНК (рис. 2А). Таким образом, все эксперименты с Cas13a-нуклеазой проводилив буфере R2. Полученные результаты показывают, что рекомбинантная Cas13a-нуклеаза может обладать рибонуклеазной активностью, которая не зависит от формирования комплексов нРНК/Cas13a. Это может быть связано с загрязнением препаратов рекомбинантной Cas13a рибонуклеазами, отличными от РНКаз А, В и С. При этом ингибитор (Murine RNase Inhibitor, https://international.neb.com/products/m0314-rnase-inhibitor-murine#Product%20Information), который подавляет активность этих ферментов, не оказывал существенного влияния на пики рРНК. Нельзя исключить наличия у рекомбинантной Cas13a неспецифической рибонуклеазной активности, например, из-за неправильного фолдинга части белковых молекул, которая существенно ниже, чем уровень коллатеральной активности (рис. 2-4). Таким образом, для однозначной детекции коллатеральной активности Cas13a-нуклеазы необходим правильный подбор её концентрации. В случае препарата Cas13a, использованного в данной работе, такой концентрацией представляется концентрация 90 нМ (12 нг/мкл). При этой концентрации взаимное наложение электрофореграмм суммарной РНК, инкубированной 1 ч в присутствии только Cas13a или в присутствии комплексов нРНК/Cas13a, с очевидностью указывает на наличие коллатеральной активности, проявляющейся в значительно более выраженном уменьшении высоты пиков рРНК во втором случае (рис. 3А). Следует отметить, что взаимное наложение электрофореграмм возможно только в том случае, если электрофоретический анализ был выполнен на одном и том же чипе, что определяется особенностями программной обработки данных биоанализатором. Также следует иметь в виду, что для другого препарата рекомбинантной Cas13a оптимальная концентрации белка может отличаться и должна быть специально подобрана, используя серию разведений препарата, как и в данной работе. Тем не менее, за начальную «точку отсчёта» могут быть приняты концентрации Cas13a, приведенных выше. Наряду с изменением высоты пиков рРНК мы наблюдали также изменение их положения на оси времени. Изменение наблюдали при повышенной концентрации Cas13a (180 нМ), но если в смеси присутствовала нРНК, то и при концентрациях Cas13a 45 и 90 нМ (рис. 1 D, 2 А и 2 B, 3 А и 3B). Присутствие РНКазы А также сказывалось на скорости электрофоретической миграции рРНК. Это может отражать влияние состава образца, формирующего стартовую зону в капилляре чипа, на наблюдаемую электрофоретическую миграцию его компонентов – явление, хорошо известное в капиллярном электрофорезе [7]. Вне зависимости от сдвига пиков рРНК на оси времени, они однозначно идентифицируются в силу их характерного появления как пары пиков, разделённых приблизительно 5-секундным интервалом, что позволяет использовать их высоту для детекции коллатеральной активности. Исходя из полученных результатов можно сформулировать основные методические рекомендации при использовании биоанализатора Agilent 2100 для тестирования коллатеральной активности CRISPR/Cas13а-нуклеазы:
К недостаткам тестирования коллатеральной активности с помощью электрофореза на чипах можно отнести отсутствие количественной оценки, а также необходимость полного заполнения микрофлюидного чипа (11 образцов) для запуска прибора. В то же время анализ каждого нового образца требует подбора оптимальной концентрации нуклеазы в серии разведений, что отчасти компенсирует последний недостаток. При этом можно отметить, что предлагаемый нами метод обходится по стоимости стандартных реактивов более чем в два раза дешевле в пересчете на одну пробу (по нашим оценкам, 4,6 долл. против 10,6 долл.) по сравнению с тестированием с помощью набора RNaseAlert® Lab Test Kit v2. Также существенно уменьшается (в 2-3 раза) расход рекомбинантного белка, необходимого для проведения тестирования вследствие существенного уменьшения требуемого объема пробы. Предлагаемый подход к тестированию рекомбинантных CRISPR/Cas-нуклеаз с коллатеральной активностью расширяет функционал биоанализаторов Agilent 2100, имеющихся сегодня в значительном числе научно-исследовательских лабораторий и биотехнологических компаний. ЗАКЛЮЧЕНИЕ Биоанализатор Agilent 2100 широко применяется в биологических и биомедицинских исследованиях и является удобным и эффективным прибором для анализа нуклеиновых кислот. Визуальное представление результатов в виде имитации электрофореза и электрофореграммы высокого разрешения помогает получить максимально полную информацию о состоянии РНК или ДНК в пробе. В данной работе мы адаптировали биоанализатор Agilent 2100 для оценки качества образцов рекомбинантной CRISPR-Cas13а-нуклеазы в лабораторных условиях как удобную альтернативу методу, основанному на расщеплении рибонуклеазой флуоресцентно-меченого синтетического РНК-субстрата. Детекция коллатеральной активности Cas13a с использованием биоанализатора Agilent 2100 представляется оправданной в случаях, когда не требуется высокая чувствительность и специфичность метода – например, при скринировании образцов рибонуклеазы, полученных в различных условиях, из различных источников или разными способами очистки. СОБЛЮДЕНИЕ ЭТИЧЕСКИХ СТАНДАРТОВ Данная работа не содержит каких-либо исследований с использованием людей и животных в качестве объектов исследования. ФИНАНСИРОВАНИЕ Работа выполнена в рамках Программы фундаментальных научных исследований в Российской Федерации на долгосрочный период (2021 - 2030 годы) № 122030100168-2. КОНФЛИКТ ИНТЕРЕСОВ/p> Авторы заявляют об отсутствии конфликта интересов. ЛИТЕРАТУРА
|