|
Конструирование и экспрессияхимерного гена реналазы человека, кодирующего n-концевую сигнальную последовательность секреторного белка пролактина
Научно-исследовательский институт, биомедицинской химии имени В.Н. Ореховича, Ключевые слова: ПЦР; клонирование; экспрессия; реналаза; пролактин; сигнальная последовательность DOI: 10.18097/BMCRM00175 ВВЕДЕНИЕ
RNLS - недавно открытый секреторный белок, который выполняет различные функции внутри и снаружи клеток [1-5]. Внутриклеточная RNLS проявляет свойства FAD-зависимой оксидоредуктазы (КФ 1.6.3.5) [5, 6], кофактор FAD которой может быть «размещён» только в полноразмерном белке, содержащем N-концевой пептид [2, 7, 8]. Одновременно этот же пептид рассматривается как сигнал внеклеточной локализации белка. Внеклеточная RNLS, лишенная N-концевого пептида, неспособна связывать FAD [8] и оказывает различные защитные эффекты на клетку посредством взаимодействия на рецепторные белки [9-11]. Механизмы и факторы, ответственные за транспорт RNLS из клетки, до конца не изучены [12]. Одним из подходов к изучению секреции RNLS из клетки и роли N-концевой сигнальной последовательности может быть создание химерных форм белка, модифицированных по N-концу. Ранее мы получили химерную RNLS человека, лишенную N-концевой последовательности (аминокислотные остатки 1-17 последовательности RNLS) [13]. В данной работе описан метод конструирования химерного гена RNLS человека, в котором N-концевая нуклеотидная последовательность гена PRL, кодирующего гормон пролактин, была вставлена вместо N-концевой нуклеотидной последовательности гена RNLS. Компьютерное моделирование с применением программ (Phobios, TargetP-2.0, SignalIP-5.0, SecretomeP 2.0a), показало, что сигнальный пептид гена PRL, соединенный с нуклеотидной последовательностью, кодирующей матричный пептид RNLS, давал классический сигнал, свойственный секреторным белкам. При конструировании химерного гена мы использовали ранее апробированный нами метод «экзоновое конструирование кодирующих последовательностей» [14], который был использован при конструировании одной из изоформ RNLS человека [15]. МАТЕРИАЛЫ И МЕТОДЫ Реактивы В работе были использованы: Tersus полимераза («Евроген», Россия), эдонуклеазы рестрикции BamHI, XhoI («Fermentas», США), Ni-сефароза («GE Healtheare», Швеция), система для очистки фрагментов ДНК (Wizard® SV Gel и PCR Clean-Up) и TA-вектор (pGEM®-T easyvector) («Promega», США), плазмидный экспрессионный вектор pET-28a(f+) и E. coli штамм Rosetta (DE3) («Novagen», Великобритания). Олигонуклеотиды для ПЦР (табл. 3) были синтезированы в «Синтол» (Россия). Остальные химические реактивы были приобретены у «Sigma-Aldrich» (США). Биоинформатический анализ Биоинформатический анализ N-концевой сигнальной последовательности RNLS, в которой возможность взаимодействия с кофактором FAD была бы минимальна при сохранении секреторного потенциала химерного белка, осуществляли с применением веб-сервисов Phobios (https://phobius.sbc.su.se/) [16], TargetP-2.0, (https://services.healthtech.dtu.dk/service.php?TargetP-2.0) [17], SignalIP-5.0 (https://services.healthtech.dtu.dk/service.php?SignalP-5.0) [18], SecretomeP 2.0a (https://services.healthtech.dtu.dk/service.php?SecretomeP-2.0) [19]. Аминокислотные последовательности химерных реналаз получали путем добавления к последовательности реналазы человека (изоформа 1) (UniProtIDQ5VYX0), у которой были удалены N-концевые 17 аминокислотных остатков (аа) (предсказанный сигнальный пептид), сигнальных пептидов внеклеточных белков человека. Длину и последовательности таких пептидов получали из соответствующих этим белкам записей в UniProt. Для полученных химерных белков была предсказана их клеточная локализация программами WolfProst (https://wolfpsort.hgc.jp/), ELSPred () [20], Hum-mPLoc2.0 (http://www.csbio.sjtu.edu.cn/bioinf/hum-multi-2/) [21], и BaCelLo (http://gpcr.biocomp.unibo.it/bacello/) [22]. Изменения во вторичных структурах полученных химерных белков оценивали с использованием программ Jpred 4 (https://www.compbio.dundee.ac.uk/jpred/) [23], psipred (http://bioinf.cs.ucl.ac.uk/psipred/) [24], GOR4 (https://npsa-prabi.ibcp.fr/cgi-bin/npsa_automat.pl?page=/NPSA/npsa_gor4.html) [25] и SOPMA (https://npsa-prabi.ibcp.fr/cgi-bin/npsa_automat.pl?page=/NPSA/npsa_sopma.html) [26]. Получение геномной ДНК человека. Геномную ДНК человека получали из лимфоцитов крови человека, полученной от здорового донора, фенол-хлороформ экстракцией, как было описано нами ранее [14, 15]. Полимеразная цепная реакция (ПЦР) ПЦР проводили в 20 мкл реакционной смеси в условиях реакции, описанной ранее [13-15], за исключением условия времени элонгации. Время элонгации при синтезе (поли) нуклеотидных фрагментов, кодирующих сигнальный пептид гена пролактина и структурный пептид (матричный пептид) RNLS, составило 60 с и 210 с соответственно. Электрофорез фрагментов ДНК Электрофорез фрагментов ДНК проводили в 2% агарозном геле с использованием трис-боратного буфера рН 8.0 [27]. Электрофорез в 12% полиакриламидном геле (ПААГ) проводили по методу Лэммли [28]. Трансформация клеток E. coli Трансформацию проводили по методу Kushner [29] с модификациями, описанными ранее [13-15]. Выделение рекомбинантного белка Выделение и очистку рекомбинантного белка осуществляли методом аффинной хроматографии с использованием Ni-сефарозы в 8 М буферном растворе мочевины, как как описано в наших предыдущих работах [14, 30]. Секвенирование ДНК Секвенирование проводили на капиллярном ДНК секвенаторе (AppliedBiosystems 310 DNA analyzer, «AppliedBiosystems», США) с использованием BigDyeTerminator v 3,1 CycleSequencingKit («Thermo Scientific», США) в соответствии с рекомендациями фирмы-производителя. Полученную последовательность ДНК соотносили с геном реналазы (NM_001031709, GeneBank), используя программу BLAST, доступную на сайте National Center for Biotechnology Information Экспрессия гена RNLS в прокариотической системе Экспрессию рекомбинантного гена в прокариотической системе (E. coli штамм DE3 Rosetta) проводили, используя ранее приведенные протоколы [13-15]. Выделение рекомбинантного белка Выделение рекомбинантного белка осуществляли методом аффинной хроматографии с использованием Ni-сефарозы в 8 М буферном растворе мочевины [15, 30].
РЕЗУЛЬТАТЫ ИОБСУЖДЕНИЕ При конструировании химерного гена RNLS, кодирующего N-концевую сигнальную последовательность секреторных белков, не связывающих FAD, были использованы: 1) - альбумин (albumin, код доступа - NM_000477 (SP - 1-18)) с сигнальным пептидом в 18 аa; 2) - пролактин (prolactin, PRL), код доступа NM_000948 с сигнальным пептидом в 28 аa; 3) - ангиотензин превращающий фермент I (ACE - angiotensin I-convertingenzyme, код доступа NM_000948 (SP 1-109)); 4) - интегрин (ITGB1 - integrinsubunitbeta 1, код доступа NM_033668) с сигнальным пептидом в 20 аa; 5) - серпин (SERPINA1 - serpin, код доступа NM_001002236 (SP - 1-24)); 6) - тромбин (thrombin, код доступа NM_000506) с сигнальным пептидом в 24 аa. Предсказание несколькими программами наличия сигнального пептида у химерных реналаз (сигнальный пептид исследуемых белков, соединенный с матричным пептидом RNLS) показало, что в большинстве случаев программы распознают наличие сигнального пептида (табл.1). Предсказание субклеточной локализации химерных белков так же указывало, что такие белки будут экскретироваться из клетки (табл.2).
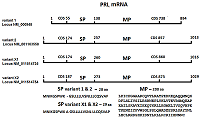
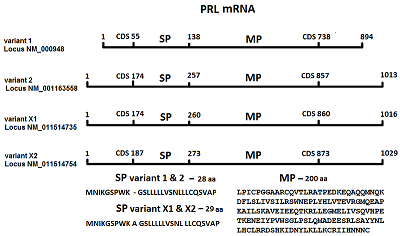
Исходя из этих данных, нами была выбрана сигнальная последовательность гена PRL. При конструировании химерного гена (сигнальная последовательность, кодируемся PRL+ матричный пептид, кодируемый RNLS) была использована ранее разработанная нами «экзоновая» стратегия клонирования генов [13, 15]. Согласно банку данных, ген пролактина (PRL) расположен на хромосоме 6 (код доступа NG_029819) и содержит 6 экзонов. Ген PRL человека имеет четыре транскрипционных варианта: вариант 1 - код доступа NM_000948; вариант 2 - код доступа NM_001163558; вариант Х1 - код доступа XM_011514753; вариант Х2 - код доступа XM_011514754 [31]. mRNA различных изомеров гена PRL имеют различную длину, но кодируют один и тот же белок с сигнальной аминокислотной последовательности (SP) 28 аа и 29 аа и одинаковой матричный пептид содержащий 200 аа. SP последовательность изомеров вариантов 1 и 2 содержит 28 аа, а изомеры варианта Х1 и Х2 содержат 29 аа, т.е. отличаются на одну аа, которая находится в SP в положении 10 (рис.1).
Нуклеотидная последовательность, кодирующая сигнальный пептид, начинается во втором и заканчивается в третьем экзоне гена PRL. Для синтеза полной сигнальной последовательности PRL методом ПЦР был синтезирован прямой праймер (SP-PRL-for) который включал: сайт рестрикции BamHI, Kozak последовательность, нуклеотидную сигнальную последовательность PRL первого экзона и начало сигнальной последовательности от второго экзона (табл. 3). Обратный праймер (SP-PRL-rev) содержал конец нуклеотидной последовательности пролактина от второго экзона и начало последовательности матричного пептида RNLS (табл. 3).
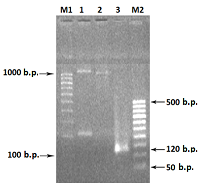
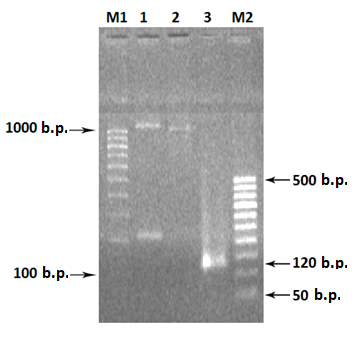
Нуклеотидную последовательность матричной части гена RNLS получали из сконструированного нами ранее вектора pET-hRenI [15]. Прямым праймером (Mat-RNLS-for) служила последовательность, комплементарная 18 нуклеотидам конца сигнальной последовательности PRL и 18 нуклеотидам начала матричной нуклеотидной последовательности RNLS. Обратный праймером (Mat-RNLS-rev) служила последовательность, включающая XhoI сайт рестрикции и 25 нуклеотидов конца матричного гена RNLS(табл. 1). Фрагмент ДНК сигнальной последовательности гена PRL получали в два этапа методом ПЦР с использованием праймеров SP-PRL-for и SP-PRL-rev. На первом этапе ПЦР синтеза сигнальной последовательности гена пролактина проводили на матрице ДНК человека, полученной из лимфоцитов крови. При анализе продукта реакции в 2% агарозном геле ампликоны визуально не обнаруживали. Для увеличения выхода ампликона проводили второй раунд амплификации, в тех же самых условиях, но с использованием в качестве ДНК-матрицы 1 мкл ампликона от первого раунда амплификации. Анализ ампликонов в 2% агарозном геле показал видимую полосу ДНК фрагмента с размером 120 пар оснований (п.о.) (рис.2, дорожка 3), что соответствует теоретическим расчетным данным (см. банк данных, например код доступа NM_000948).
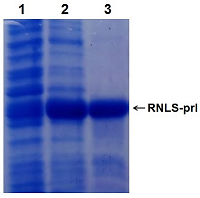
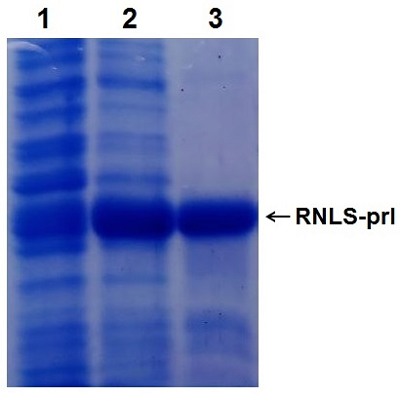
Синтез матричной ДНК гена RNLS проводили методом ПЦР с использованием праймеров Mat-RNLS-for и Mat-RNLS-rev (табл. 3). В качестве матрицы ДНК использовали вектор pET-hRenI, полученный нами ранее. В результате анализа продукта ПЦР-реакции в 2% агарозном геле явно выявлялась полоса в 990 п.о., которая соответствует расчетным данным (рис.2, дорожка 2). При получении химерной ДНК, состоящей из сигнальной последовательности PRL и матричной последовательности RNLS, в ПЦР-реакции использовали праймеры SP-PRL-for и Mat-RNLS-rev, фланкирующие концы которых содержали сайты рестрикции BamHI и XhoI соответственно. В качестве ДНК-матрицы использовали по 1 мкл ампликонов от синтеза ДНК сигнальной последовательности пролактина и матричной последовательности RNLS. В результате ПЦР реакции был получен фрагмент ДНК в 1092 п.о., который соответствовал расчетным данным (рис. 1, дорожка 1). Полученный химерный фрагмент ДНК после ПЦР реакции очищали согласно протоколу производителя с помощью системы очистки фрагментов ДНК («Wizard»). Очищенный таким образом фрагмент ДНК встраивали в вектор pGEM®-T Easy и трансформировали им клетки E.coli BL-21 (DE3). Плазмидные ДНК из трансформированных клонов подвергали рестрикционному анализу по сайтам BamHI/XhoI. Векторы, содержащие встроенный фрагмент ДНК в области 1100 п.о., были секвенированы. Соответствие нуклеотидной последовательности анализировали с сигнальной последовательностью гена PRL человека (код доступа NM_000948) и нуклеотидной последовательностью матричного пептида RNLS (код доступа NM_001031709). Анализ показал, что кодирующая последовательность химерного гена RNLS соответствовала сигнальной нуклеотидной последовательности изомеров вариантов 1 и 2 гена PRL и состояла из 84 нуклеотидов и матричной нуклеотидной последовательности гена RNLS сконструированного нами вектора pET-hRenI. Таким образом, выбранная нами стратегия получения кодирующих последовательностей гена является приемлемой при конструировании химерных генов [31]. Далее, фрагмент ДНК 1100 п.о. встраивали по сайтам BamHI/XhoI в вектор pET-28a (+) и затем трансформировали клетки E. coli. В результате проведенного клонирования с последующим рестрикным анализом и секвенированием был получен клон, содержащий матричный ген RNLS с сигнальной последовательностью гена PRL, обозначенный как pET-PRL-RenI. Экспрессию вектора pET-PRL-RenI проводили в прокариотической системе в клетках E. coli (штамм DE3 Rosetta), которая давала хороший выход целевого белка. Очистку рекомбинантного белка проводили методом хроматографии на Ni-Sepharose[30]. После очистки был получен рекомбинантный белок(RNLS-prl) с расчетной молекулярной массой 42.9кДа (рис.3).
ЗАКЛЮЧЕНИЕ В данной работе сконструирована химерная генетическая конструкция, кодирующая RNLS человека, у которой «родная» сигнальная последовательность, участвующая в связывании кофактора FAD, а также секреции этого белка из клетки, была заменена на N-концевую сигнальную последовательность секреторного белка пролактина. Эта генетическая конструкция была экспрессирована в клетках E. coli (штамм DE3 Rosetta) с хорошим выходом целевого белка. СОБЛЮДЕНИЕ ЭТИЧЕСКИХ СТАНДАРТОВ Здоровые добровольцы дали информированное согласие на использование их образцов крови в исследовательских целях. ФИНАНСИРОВАНИЕ Исследования выполнены в рамках проекта РФФИ № 20–015–00104. ЛИТЕРАТУРА
|