Использование праймеров с высокой степенью мечения в полимеразной цепной реакции
1Научно-исследовательский институт биомедицинской химии имени В. Н. Ореховича,
119121, Москва, ул. Погодинская, 10; e-mail: konstantin157@yandex.ru
2Химический факультет Московского государственного унивеситета им. М.В.Ломоносова,
119991, Москва, Ленинские горы, 1/3
Ключевые слова: праймеры; флуоресцеин; ПЦР
DOI:10.18097/BMCRM00194
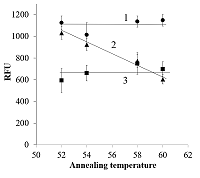
Флуоресцентно-меченую ДНК широко используют в различных биоаналитических приложениях. В ряде случаев высокий уровень меченья ДНК является преимуществом. Одним из способов получения фрагментов ДНК, несущих множественные флуоресцентные метки, является использование полимеразной цепной реакции (ПЦР) с праймерами с высокой степенью мечения. В данной работе мы исследовали, как присутствие многочисленных молекул флуоресцеина, прикреплённых к праймеру, влияет на результат ПЦР. Было обнаружено, что расположение флуоресцеиновой метки на 3′-конце праймера или вблизи него может ингибировать амплификацию (вплоть до полной остановки, когда метка находится непосредственно на 3′-концевом или соседнем с ним нуклеотиде). Механизм, посредством которого присутствие флуоресцеиновых меток на 3’-конце праймера или вблизи него влияет на эффективность ПЦР, довольно неоднозначен и может включать как стерическое препятствование связыванию полимеразы прикреплённой молекулой флуоресцеина, так и дестабилизацию дуплекса праймер-матрица. Тем не менее, если множественные флуоресцеиновые метки прикреплены к нуклеотидам праймера таким образом, что по меньшей мере три нуклеотида с его 3′-конца остаются немодифицированными, то получение фрагментов ДНК с высоким уровнем мечения возможно с помощью ПЦР, хотя и с заметно меньшим выходом, чем при использовании немодифицированных праймеров.

|
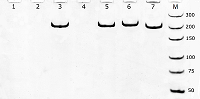
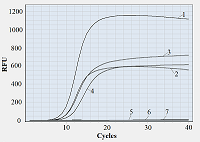
Рисунок 1.
Характерные амплификационные кривые. Пары праймеров: 1 – F/R, 2 – F/R*(4),
3 – F*(6)/R, 4 – F*(6)/R*(4), 5 – F*(1)/R, 6 – F*(2)/R, 7 – F/R*(1). |
|
ЗАКРЫТЬ

|
Таблица 1.
Последовательности используемых праймеров. Последовательности показаны
с 5’- к 3’-концу. Звездочка в последовательности праймеров после “t” обозначает тимидин с присоединенным флуоресцеином (FAM), а в названии праймера – праймер с флуоресцеиновыми метками. Число в круглых скобках в названии праймера указывает положение последнего модифицированного тимидина с 3’-конца праймера. |
ФИНАНСИРОВАНИЕ
Работа выполнена в рамках Программы фундаментальных научных исследований в Российской Федерации на долгосрочный период (2021 - 2030 годы) (№ 122030100170-5).
- Faltin B., Zengerle R., von Stetten F. (2013) Current methods for fluorescence-based universal sequence-dependent detection of nucleic acids in homogenous assays and clinical applications. Clinical Chemistry, 59(11), 1567-1582. DOI
- Klöcker N., Weissenboeck F.P., Rentmeister A. (2020) Covalent labeling of nucleic acids. Chemical Society Reviews, 49(23), 8749-8773. DOI
- Sharma R., Meister P. (2020) Generation of Inexpensive, Highly Labeled Probes for Fluorescence In Situ Hybridization (FISH). STAR Protocols, 1(1), 100006. DOI
- Anderson J.P., Angerer B., Loeb L.A. (2005) Incorporation of reporter-labeled nucleotides by DNA polymerases. Biotechniques, 38(2), 257-264. DOI
- Volkova O.S., Chudinov A.V., Lapa S.A. (2021) Study of the multiple incorporation of modified nucleotides into the growing DNA strand. Fine Chemical Technologies, 16(2), 148-155. DOI
- 6. Jun Y.W., Harcourt E.M., Xiao L., Wilson D.L., Kool E.T. (2022) Efficient DNA fluorescence labeling via base excision trapping. Nature Communications, 13(1), 5043. DOI
- Panattoni A., Pohl R., Hocek M. (2018) Flexible Alkyne-Linked Thymidine Phosphoramidites and Triphosphates for Chemical or Polymerase Synthesis and Fast Postsynthetic DNA Functionalization through Copper-Catalyzed Alkyne-Azide 1,3-Dipolar Cycloaddition. Organic Letters, 20(13), 3962-3965. DOI
- Sudarsan S., Prabhu A., Prasad D., Mani N.K. (2023) DNA compaction enhances the sensitivity of fluorescence-based nucleic acid assays: a game changer in point of care sensors? Analyst, 148(10), 2295-2307. DOI
- Suprun E.V., Khmeleva S.A, Kutdusova G.R, Duskaev I.F., Kuznetsova V.E., Lapa S.A., Chudinov A.V., Radko S.P. (2020) Deoxyuridine triphosphates modified with tyrosine or tryptophan aromatic groups for direct electrochemical detection of double-stranded DNA. Electrochimica Acta, 362, 137105. DOI
- Stadhouders R., Pas S.D., Anber J., Voermans J., Mes T.H., Schutten M. (2010) The effect of primer-template mismatches on the detection and quantification of nucleic acids using the 5' nuclease assay. The Journal of Molecular Diagnostics, 12(1), 109-117. DOI
- Chudinov A.V., Kuznetsov V.E., Shershov V.E., Spitsyn M.A., Guseinov T.O., Lapa S.A., Timofeev E.N., Zasedatelev A.S., Kiseleva Y.Y., Archakov A.I., Lisitsa A.V., Radko S.P. (2017) Structural and functional analysis of biopolymers and their complexes: enzymatic synthesis of high-modified DNA. Molecular Biology, 51(3), 474-482. DOI
- Kwok S., Kellogg D.E., McKinney N., Spasic D., Goda L., Levenson C., Sninsky J.J. (1990) Effects of primer-template mismatches on the polymerase chain reaction: human immunodeficiency virus type 1 model studies. Nucleic Acids Research, 18(4), 999-1005. DOI
- Huang M.M., Arnheim N., Goodman M.F. (1992) Extension of base mispairs by Taq DNA polymerase: implications for single nucleotide discrimination in PCR. Nucleic Acids Research, 20(17), 4567-4573. DOI