Идентификация рибосомных футпринтов на электрофоретическом геле при профилировании транслатома: использование ДНК-маркёров
Научно-исследовательский институт биомедицинской химии имени В. Н. Ореховича,
119121, Москва, ул. Погодинская, 10; e-mail: khmelevaswetlana@yandex.ru
Ключевые слова: профилирование транслатома, рибосомные футпринты, выделение, ДНК маркёры
DOI:10.18097/BMCRM00195
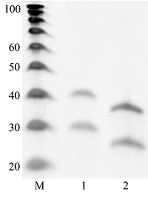
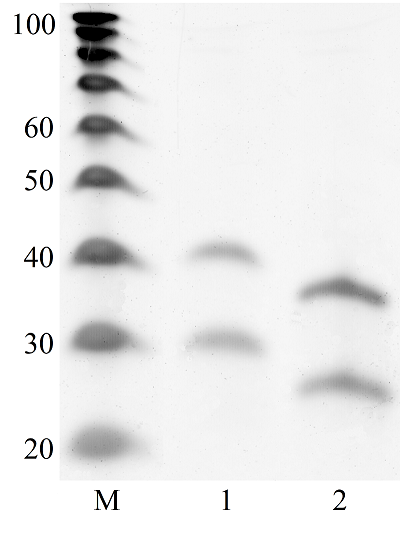
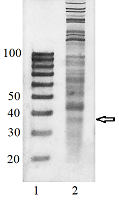
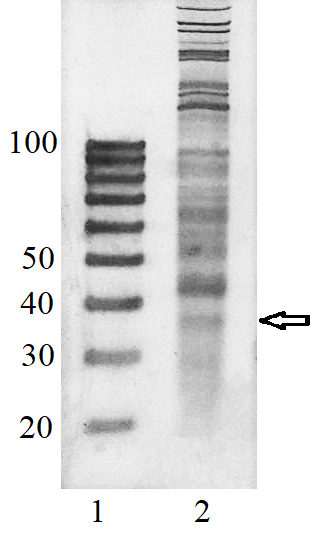
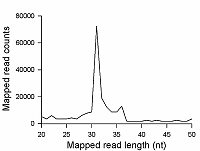
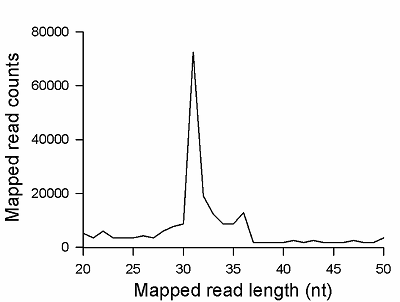
Коммерческие ДНК-маркёры тестировались как замена РНК-стандартов длины молекулы для идентификации рибосомных футпринтов (фрагментов РНК длиной около 30 нуклеотидов) на электрофоретическом полиакриламидном геле при профилировании транслатома. Было обнаружено, что синтетические РНК-олигонуклеотиды длиной 25 и 35 нуклеотидов мигрируют медленнее, чем синтетические ДНК-олигонуклеотиды соответствующей длины и последовательностей, и их положение на геле совпадает с положением ДНК-олигонуклеотидов длиной 30 и 40 нуклеотидов, соответственно, из коммерческого набора стандартов длин ДНК-олигонуклеотидов «IDT 20/100 DNA oligo length standards». Используя данный набор и РНК, выделенную из препарата, обогащенного рибосомами (полученного фракционированием на колонках MicroSpin S-400 клеточного лизата HepG2, обработанного РНКазой I), можно идентифицировать положение полосы предполагаемых рибосомных футпринтов на геле, что было подтверждено измерением длины фрагментов РНК, выделенных из полосы, при проведении их секвенирования методом RNA-seq.
ФИНАНСИРОВАНИЕ
Работа выполнена в рамках Программы фундаментальных научных исследований в Российской Федерации на долгосрочный период (2021 - 2030 годы) (№ 122030100170-5).
ЛИТЕРАТУРА
- Ingolia N.T., Ghaemmaghami S., Newman J.R., Weissman J.S. (2009) Genome-wide analysis in vivo of translation with nucleotide resolution using ribosome profiling. Science, 324(5924), 218-223. DOI
- Wang Y., Zhang H., Lu J. (2020) Recent advances in ribosome profiling for deciphering translational regulation. Methods, 176, 46-54. DOI
- McGlincy N.J., Ingolia N.T. (2017) Transcriptome-wide measurement of translation by ribosome profiling. Methods, 126, 112-129. DOI
- Chen H., Meisburger S.P., Pabit S.A., Sutton J.L., Webb W.W., Pollack L. (2012) Ionic strength-dependent persistence lengths of single-stranded RNA and DNA. The Proceedings of the National Academy of Sciences of the U S A, 109(3), 799-804. DOI
- Bao L., Zhang X., Jin L., Tan Z.J. (2016) Flexibility of nucleic acids: From DNA to RNA. Chinese Physics B, 25, 018703. DOI
- Deinichenko K.A., Krasnov G.S., Radko S.P., Ptitsyn K.G., Shapovalova V.V., Timoshenko O.S., Khmeleva S.A., Kurbatov L.K., Kiseleva Y.Y., Ilgisonis E.V., Pyatnitskiy M.A., Poverennaya E.V., Kiseleva O.I., Vakhrushev I.V., Tsvetkova A.V., Buromski I.V., Markin S.S., Zgoda V.G., Archakov A.I., Lisitsa A.V., Ponomarenko E.A. (2021) Human CHR18: “Stakhanovite” Genes, Missing and uPE1 Proteins in Liver Tissue and HepG2 Cells. Biomedical Chemistry: Research and Methods, 4(1), e00144. DOI
- Wang H., Wang Y., Yang J., Zhao Q., Tang N., Chen C., Li H., Cheng C., Xie M., Yang Y., Xie Z. (2021) Tissue- and stage-specific landscape of the mouse translatome. Nucleic Acids Research, 49(11), 6165-6180. DOI
- Langmead, B., Trapnell, C., Pop, M., Salzberg, S.L. (2009) Ultrafast and memory-efficient alignment of short DNA sequences to the human genome. Genome Biology 10, R25. DOI