|
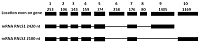
Получение C-концевых последовательностей реналазы-1 и реналазы-2 человека, кодируемых альтернативными экзонами Научно-исследовательский институт биомедицинской химии имени В.Н. Ореховича, 119121 Москва, Погодинская ул., д.10; *e-mail: valfed38@yandex.ru Ключевые слова: реналаза (RNLS); альтернативные экзоны гена RNLS; изоформы реналазы RNLS1 и RNLS2; клонирование; экспрессия; очистка белка DOI: 10.18097/BMCRM00228 ВВЕДЕНИЕ Реналаза (RNLS) – открытый в 2005 г. белок, который играет разные роли внутри и снаружи клеток [1-5]. Внутриклеточная RNLS представляет собой FAD-зависимую оксидоредуктазу (КФ 1.6.3.5) [5, 6], которая осуществляет окисление изомерных форм b-NAD(P)H, восстановленных по 2 или 6 положению никотинамидного кольца вместо метаболически активного 4 положения [7]. Внеклеточная RNLS, лишенная своего N-концевого пептида и кофактора FAD, проявляет различные защитные эффекты при помощи некаталитических механизмов [8-11]. Детекция RNLS в крови свидетельствует в пользу того, что этот белок может дистантно действовать на клетки различных органов [11]. Основными формами RNLS человека служат RNLS1 и RNLS2 [12, 13], которые отличаются С-концевой аминокислотной последовательностью, кодируемой альтернативными экзонами (рис. 1). Поскольку обе RNLS реагируют с антителами к RNLS1 [12], это серьезно осложняет их анализ в организме в норме и при патологии.
Одним из подходов к решению данной проблемы может быть создание антител к изоформ специфичным С-концевым аминокислотным последовательностям RNLS1 и RNLS2, кодируемым альтернативными экзонами. В данной работе мы описываем метод получения С-концевых аминокислотных последовательностей RNLS1 и RNLS2, слитых с дигидрофолатредуктазой – белком-ферментом, часто используемым для формирования слитых (т.н. фьюжн) пептидных последовательностей для наработки и последующего иммунологического применения коротких пептидов [14]. МЕТОДИКА Реактивы В работе использованы следующие реагенты: Набор Tersus-ДНК полимераза («Евроген», Россия), эдонуклеазы рестрикции BglII, PstI, ДНК маркеры («Ферментас», Латвия), маркеры молекулярной массы белка («BOI-RAD», США). Ni-сефароза («GE Healtheare», Швеция), система для очистки фрагментов ДНК Wizard® SVGel и PCR Clean-Up («Promega», США), вектор pGEM-T(«Promega», США), вектор pQE-40 («Novagen», Великобритания). Олигонуклеотиды для ПЦР были синтезированы по запросу «Евроген» (табл. 1). E. coli штамм Rosetta (DE3) получен из «Novagen». Остальные реактивы приобретены в «Sigma-Aldrich» (США). Метод полимеразной цепной реакции (ПЦР) Реакцию проводили в индивидуальных пробирках «Eppendorf» для ПЦР реакции емкостью 500 мкл в объеме 20 мкл. Реакционная смесь содержала: Tersus ПЦР буфер, 0.2 мМ dNTP; прямой и обратный праймеры по 4 мкМ, 50 нг матричной ДНК и 0.5 ед. Tersus-ДНК-полимеразы. Синтез RNLS1-9ex и RNLS2-10ex осуществляли на матрице ДНК вектора pET-RenI и вектора pET-RenII соответственно, которые были получены нами ранее [12, 15]. Условия ПЦР реакции были следующие: денатурация – 95ºС в течение 3 мин – 1 цикл; синтез ДНК ампликонов – 92ºС – 20 с, 55ºС – 15 с, 72ºС – от 60 с – 30 циклов; заключительный цикл 72ºС – 3 мин. Электрофорез фрагментов ДНК Электрофорез фрагментов ДНК проводили в 2% агарозном геле с использованием трис-боратного буфера рН 8.0 [16]. Секвенирование ДНК Секвенирование проведено по запросу в фирме «Евроген». Полученную последовательность ДНК соотносили с геном реналазы (NM_001031709 и NM_018363, GeneBank), используя программу BLAST, доступную на сайте Nationa Center for Biotechnology Information (http://www.ncbi.nlm.nih.gov). Трансформация клеток E. coli Трансформацию проводили по методу Kushner с модификациями, описанными ранее [12, 15, 17]. Выделение рекомбинантного белков, кодируемых RNLS1-9ex и RNLS2-10ex Выделение и очистку рекомбинантного белка осуществляли методом аффинной хроматографии с использованием Ni-сефарозы в 8 М буферном растворе мочевины, как описано в наших предыдущих работах [12, 15, 17, 18]. Электрофорез белков в ПААГ Электрофорез белков проводили в 12% полиакриламидном геле (ПААГ) по методу Лэммли [19]. РЕЗУЛЬТАТЫ Для клонирования альтернативных экзонов RNLS1-9ex и RNLS2-10ex гена RNLS в вектор pQE40 по сайтам рестрикции BglII и PstI было синтезировано 2 пары прймеров. Эти экзоны кодируют аминокислотные последовательности, определяющие структурные различия RNLS1 и RNLS2. Нуклеотидные последовательности этих праймеров приведены в таблице 1. Прямые праймеры (Re1-9ex-for и Re2-10ex-for) содержали сайт рестрикции BglII. Обратные праймеры Re1-9ex-rev и Re2-10ex-rev – сайт рестрикции PstI (табл. 1).
При синтезе RNLS1-9ex методом ПЦР использовали прямой праймер Re1-9ex-for и обратный праймер Re1-9ex-rev. Матрицей ДНК служила плазмидный вектрор pET-RenI. ПЦР реакцию проводили по протоколу, который был нами ранее отработан [12, 15]. В результате ПЦР реакции получали ДНК ампликон с расчетным размером147 п.о. (рис. 2 дорожка 1).
После электрофореза в 2% агарозном геле ДНК ампликоны RNLS1-9ex и RNLS2-10ex очищали системой Wizard (система очистки фрагментов ДНК из агарозного геля), следуя протоколу производителя. Очищенные ДНК ампликоны RNLS1-9ex и RNLS2-10ex клонировали в плазмидный вектор в pGEM-T. Процедура клонирования, отбор клонов и секвенирование подробно было описано нами ранее [12, 15]. Секвенированные последовательности RNLS1-9ex и RNLS2-10ex сравнивали с последовательностями из базы данных RNLS1 (NM_001031709.2) и RNLS2 (NM_018363.3) методом BLAST анализа [12, 15, 17]. ДНК, содержащие нуклеотидные последовательности RNLS1-9ex и RNLS2-10ex в векторе pGEM-T, вырезали по сайтам рестрикции BglII/PstI и переклонировали по этим же сайтам в вектор pQE40. Вектор pQE40 был выбран потому, что, во-первых, он кодирует перед N-концом 6хHis участок, необходимый для очистки рекомбинантного белка и, во-вторых, содержит нуклеотидную последовательность, кодирующую белок дигидрофолатредуктазу (dihydrofolatereductase; DHFR) с молекулярной массой около 20 кДА. Этот белок обычно используют в качестве т.н. добавочного фьюжн белка при экспрессии полипептидов (рис. 3) [14].
В результате клонирования RNLS1-9ex и RNLS2-10ex в плазмидный вектор pQE40 были получены два экспрессионных вектора, обозначены как pQE-ReI-9ex и pQE-ReII-10ex, соответственно. Экспрессия гена RNLS в прокариотической системе Экспрессию рекомбинантнных генов в плазмидах pQE-ReI-9ex и pQE-ReII-10ex в E. coli штамм Rosetta проводили по протоколу, используемому ранее [12, 15]. В результате клонирования были отобраны по восемь клонов, содержащих экспрессионные вектора. При контрольной экспрессии рекомбинантного белка в клетках E. coli с индукцией ИПТГ, содержащего вектор pQE-ReI-9ex, был отобран клон №5, содержащий максимальный экспрессионный белок ReI-9ex в области 28.5 kDa (рис 4 А, дорожка 5). При контрольной экспрессии вектора pQE-ReII-10ex клетках E. coli был отобран клон №3, содержащий максимальный экспрессионный белок ReII-10ex в области 25.6 kDa (рис 4-В, дорожка 3). Отобранные клоны №3 и №5, содержащие максимально экспрессируемый белок ReI-9ex и ReII-10ex соответственно, были использованы для наработки и очистки рекобинантного белка.
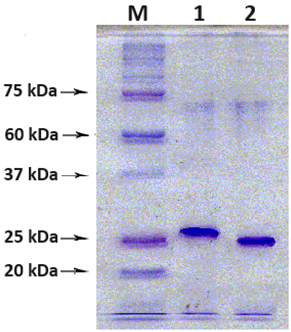
Выделение рекомбинантного белка Очистку рекомбинантного белка ReI-9ex и ReII-10ex осуществляли методом аффинной хроматографии с использованием Ni-сефарозы в 8 М буферном растворе мочевины, как было описано ранее в наших работах [12, 15, 17, 18]. Полученные в результате хроматографической очистки препараты белка анализировали электрофоретически в 12% ПААГ (рис. 5). После очистки были получены рекомбинантные белки ReI-9ex и ReII-10ex с высокой долей очистки (около 95% чистоты), которые будут использованы в дальнейших исследованиях.
ОБСУЖДЕНИЕ В результате клонирования экзонов RNLS1-9ex и RNLS2-10ex в pQE40 вектор были получены экспрессионные векторы pQE-ReI-9ex и pQE-ReII-10ex, в которых генетические конструкции ReI-9ex и ReII-10ex были слиты с DHFR. В результате их экспрессии в клетках E. coli штамм Rosetta, были получены рекомбинантные белки ReI-9ex и ReII-10ex с молекулярными массами 28.5 кДа и 25.6 кДа, соответствующие молекулярным массам целевых белков. В результате хроматографической очистки на колонке, содержащей Ni‑сефарозу, были получены высокоочищенные препараты реомбинантных белков ReI-9ex и ReII-10ex с электрофоретической чистотой около 95%. При использовании этих белков для получения антител против C-концевых последовательностей RNLS1 и RNLS2 человека, следует помнить, что уровень RNLS в крови человека, по данным разных авторов (см. например, таблицу 1 в [20]), варьирует от 20 нг/мл до 4260 нг/мг. С одной стороны, это существенно выше уровня DHFR (<10 нг/мл [21]). С другой – возможное взаимодействие DHFR с антителами к так называемым фьюжн белкам можно скорректировать предварительной обработкой анализируемых биологических жидкостей коммерчески доступными препаратами антителами к DHFR. СОБЛЮДЕНИЕ ЭТИЧЕСКИХ СТАНДАРТОВ Данная работа не включает исследования, в которых в качестве объекта выступали люди или животные. ФИНАНСИРОВАНИЕ Работа выполнена в рамках Программы фундаментальных научных исследований в Российской Федерации на долгосрочный период (2021–2030 гг.) (№ 122030100170-5). КОНФЛИКТ ИНТЕРЕСОВ Авторы заявляют об отсутствии конфликта интересов. ЛИТЕРАТУРА
|