|
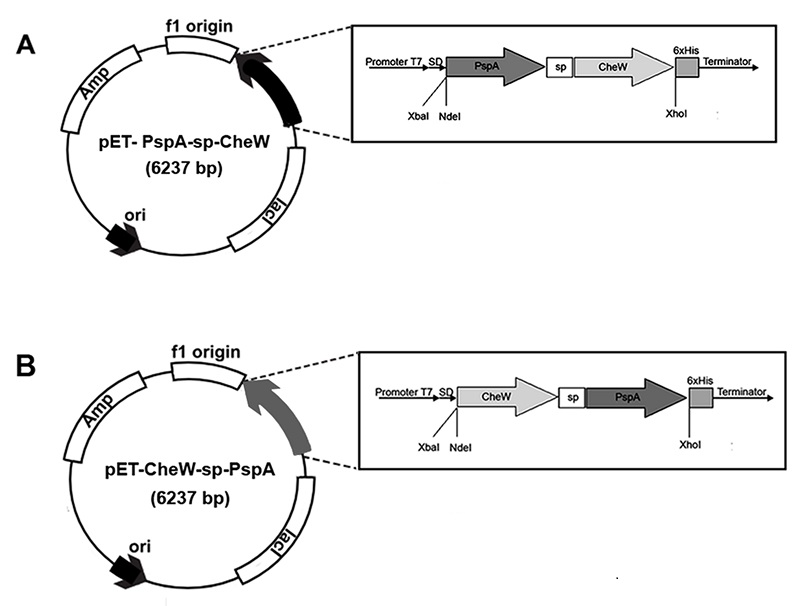
Рекомбинантные белки, объединяющие междоменный регион пневмококкового поверхностного антигена “А” и адаптивный полипептид W-типа бактерий рода Thermotoga, в качестве потенциальной компонентной базы для разработки новых диагностикумов и генно-инженерных субъединичных вакцин против пневмококковой инфекции 1Научно-исследовательский институт биомедицинской химии им. В.Н. Ореховича, 119121, Москва, Погодинская ул., 10; *e-mail: molbiol_ibm@inbox.ru Ключевые слова: химерные белки; поверхностный антиген пневмококков; термостабильный белок; вакцинно-ценный белок; Escherichia coli; гетерологическая экспрессия DOI: 10.18097/BMCRM00237 ВВЕДЕНИЕ Мезофильный микроорганизм Streptococcus pneumoniae поражает верхние дыхательные пути и является основной причиной таких опасных заболеваний, как менингит, бактериемия, острый отит, синусит и пневмония [1]. Пневмококковые инфекции по-прежнему ежегодно уносят миллионы жизней, особенно в развивающихся странах [2]. Современные антипневмококковые вакцины основаны на капсульных полисахаридах. Между тем, возрастающая устойчивость клинических изолятов S. pneumoniae к антибиотикам определяет актуальность разработки новых и повышения эффективности уже существующих стрептококковых вакцин. Вакцины, состоящие из полисахаридов пневмококка, эффективны в основном только в отношении конкретных серотипов; при этом само производство является сложным и дорогостоящим процессом, что ограничивает их широкое использование [3]. В связи с этим усилия современной иммунологической науки сосредоточены на характеристике белковых антигенов S. pneumoniae, которые могли бы обеспечить более широкую защиту на уровне носоглоточного носительства и бактериемии [4-6]. Одним из таких перспективных полипептидов является поверхностный антиген пневмококка А (PsaA) массой 37 кДа, представляющий собой основной фактор вирулентности данной бактерии, который генетически консервативен среди большинства серотипов S. pneumoniae [7, 8]. Ряд исследований показал, что активная и пассивная иммунизация полноразмерным PspA способны обеспечить защиту лабораторных животных от летального исхода при заражении пневмококком [9-11]. Усечённые фрагменты PspA были способны обеспечить защиту, но только в присутствии полного адъюванта Фрейнда (CFA). В отсутствие CFA фрагменты были менее иммуногенными, чем нативный PspA [12]. Помимо этого, следует отметить, что современные вакцины на основе этого антигена индуцируют антитела с низкой перекрёстной реактивностью, что приводит к ограниченному охвату серотипов патогена [13]. Выделение нативного полноразмерного PspA из экстрактов клеточных стенок пневмококков сильно затруднено тем фактом, что он интенсивно агрегирует. Использование стандартных сорбентов в данном случае малоэффективно, поэтому для подобных целей используют специфические дорогостоящие сорбенты, основанные на модифицированной холином сефарозе, что значительно удорожает конечный продукт [14]. Рекомбинантные варианты PspA не стабильно продуцируются в стандартных экспрессионных системах и по-прежнему требуют использования дорогостоящей многостадийной очистки препарата от эндотоксинов и белков продуцента, включающей обработку катионными детергентами, анионообменную хроматографию, криопреципитацию, катионообменную и мультимодальную хроматографию [13]. Различные направления генной и белковой инженерии предоставляют возможность реализации новых индивидуальных стратегий в соответствии с требованиями конкретного технологического процесса. Одним из таких направлений является технология химеризации белков, иначе называемая fusion-технологией, при которой разные системы слияния (протеазы, иммуноглобулины, карбогидратсвязывающие модули, стрессовые белки и т.д.) используются для решения различных технологических целей и задач: для повышения иммуногенности полипептида, улучшения гетерологической экспрессии и растворимости рекомбинантного белка, оптимизации его очистки от клеточного дебриса и т.п. [15-17]. Таким образом, задачей данного исследования стала разработка на базе технологии слитных генно-инженерных конструкций необходимой компонентной базы для оптимизации антипневмококковых субъединичных вакцин на примере модельного объекта, в качестве которого выступил инвариантный для различных биоваров междоменный участок (BP) PspA, захватывающий часть наиболее иммуногенного таксон-специфического региона (CDR) и часть пролин-богатого домена (PRD). В качестве потенциальной системы слияния, которая может быть использована для получения белка интереса в растворимом виде и оптимизации его тонкой очистки, был предложен адаптивный полипептид W-типа (CheW) из термостабильных бактерий рода Thermotoga [18]. Подобный выбор был продиктован тем, что некоторые белки, ответственные за хемотаксис, помимо транспортных функций могут участвовать в фолдинге белков и их защите от стресса. Другими словами, данные белки способны проявлять шапероноподобные свойства [19], тем самым влияя на биосинтез белка, что актуализирует изучаемую проблему. Полученные в ходе работы результаты расширяют представления о функционировании подобных систем и могут стать толчком к разработке новых специализированных инструментов для производства иммунологически ценных белков в клетках E. coli. МЕТОДИКА Анализ данных Последовательности, аннотированные в международной базе данных GenBank, были проанализированы с использованием базового инструмента по поиску и локальному выравниванию («BLAST», США) [20]. Для множественного и попарного выравнивания биоинформатических последовательностей, а также для последующей визуализации сайтов с различной гомологией были использованы программы DNASIS v 2.5 («Hitachi Software Engineering Co., Ltd.», Япония) и Clustal W [21]. Моделирование генно-инженерных конструкций и картирование рекомбинантных плазмид проводили с использованием программы Clone Manager 4.0 («Scientific & Educational Software», США). Бактериальные штаммы В работе использовали штамм Escherichia coli BL21(DE3) pLysS E. coli str. B F– ompT gal dcm lon hsdSB(rB–mB–) λ(DE3 [lacI lacUV5-T7p07 ind1 sam7 nin5]) [malB+]K-12(λS) pLysS[T7p20 orip15A](CmR) и штамм XL1-Blue [recA1 endA1 gyrA96 thi-1 hsdR17 supE44 relA1 lac [F' proAB lacIqZdeltaM15 Tn10(TetR)]] («Stratagene», США). ДНК и трансформация клеток В работе были использованы плазмидный вектор pET21a («Novagen», США) и синтетические олигонуклеотиды, синтезированные твердофазным амидофосфитным методом в «Евроген» (Россия). Компетентные клетки E. coli трансформировали, как описано в [22]. Выделение и элюция ДНК Выделение плазмидной ДНК из бактериальных клеток осуществляли стандартным методом щелочного лизиса с использованием додецилсульфата натрия (ДСН) [23]. Элюцию фрагментов ДНК из агарозного геля проводили с помощью набора «Cleanup S-Cap» («Евроген») в соответствии с рекомендациями производителя. Конструирование рекомбинантной ДНК При конструировании за основу брали аннотированные в GenBank нативные биоинформатические последовательности пневмококкового антигена PspA (GenBank: AAC62252.1) и термостабильного адаптивного полипептида W-типа (CheW) из бактерий T. petrophila (GenBank: ABQ46258.1). Генно-инженерные конструкции собирали из синтетических олигонуклеотидов, как описано в [24]. При этом использовался мастер-микс Gibson Assembly Cloning Kit (ISO буфер; Phusion ДНК-полимераза; Taq ДНК лигаза; T5-экзонуклеаза) («NEB», США). Правильность сборки кодирующих генов подтверждали рестрикционным картированием и ДНК-секвенированием. Секвенирование молекул ДНК с использованием меченных терминаторов и разделение продуктов секвенирования проведено на автоматическом секвенаторе ABI PRISM 3730xl («Applied Biosystems», США) в компании «Евроген». Анализ сложных хроматограмм секвенирования осуществляли в программе Chromas 2.6.6 («Technelysium Pty Ltd.», Australia). Полимеразная цепная реакция ПЦР проводили на приборе Терцик («ДНК-технология», Россия). Реакционная смесь (25 мкл) содержала: 2.5 мкл 1X Phusion буфера («NEB»), dNTP в конечной концентрации 400 мкМ, 5x10-7 M каждого праймера, 1 мкл Phusion ДНК полимеразы («NEB») до 5 Единиц активности фермента на объём пробы и необходимое количество ДНК матрицы. Параметры амплификации: Предварительная денатурация: 98°C, 30 с; Шаг 1 (денатурация): 98°C, 10 с; Шаг 2 (отжиг): 59-65°C, 30 с; Шаг 3 (элонгация): 72°C, variable; Шаги с 1 по 3 необходимо повторить 30 раз с целью амплификации достаточного количества ДНК-мишени для последующей визуализации на геле. Финальная элонгация: 72°C, 5 мин; Хранение: 10°C. Режим амплификации устанавливали точный. Культивирование штамма-продуцента Культуру E. coli BL21(DE3) pLysS, продуцирующую рекомбинантный белок, выращивали на среде LB в присутствии ампициллина. Трансформанты пересевали в разведении 1:50–1:100 на свежую среду LB и дальнейшее выращивание проводили при 37°С и интенсивном перемешивании при 250 об/мин на шейкере-инкубаторе ES-20 («Biosan», Латвия). Индуктор изопропил-β-D-1-тиогалактопиранозида (ИПТГ) добавляли до финальной концентрации 0.5 мМ после достижения суспензией клеток значений оптической плотности OD600 = 0.7–0.9 ед. Предварительно отбирали необходимые контрольные пробы. Далее клетки культивировали на шейкере-инкубаторе еще 16–18 ч при 27°С. Термолизис Осадки индуцированных клеток E. coli суспендировали в буфере «Т» (20 мМ Tris-HCl, 50 мкМ NaCl, 1 мкМ EDTA, 0.1% Triton X-100, pH (5.7-8.5)) из расчёта 10% от объёма культуры. Суспензию клеток обрабатывали ультразвуком на приборе Scientz JY96-IIN («Drawell», Китай) в жёстком режиме (22 кГц, 2-3 раза по 50 с с интервалом в 20 с) на льду. Пробы прогревали при 75-99°С в течение 40-45 мин, после чего клеточный дебрис и денатурировавшие белки удаляли центрифугированием. Молекулярную массу, чистоту рекомбинантных белков в супернатанте и растворимость определяли электрофорезом в 12%-ном ДСН-ПААГ. Маркером для сравнительной идентификации молекулярного веса белков служил Low Range Protein Ladder (LRPL) («Thermo Fisher Scientific», США). РЕЗУЛЬТАТЫ И ОБСУЖДЕНИЕ Биоинформатическое планирование Запланированный химерный белок должен объединять иммуногенный B-регион поверхностного антигена из S. pneumoniae (PspA) (GenBank: AAC62252.1) и адаптивный полипептид CheW анаэробных гипертермофильных бактерий T. petrophila (GenBank: ABQ46258.1) в N- и С-концевых вариациях относительно термостабильного белка. Для пространственного разделения разнофункциональных доменов между ними был запланирован инертный глицин-сериновый линкер, который не накладывает ограничений на конформацию или взаимодействия связанных между собой белковых партнёров [25]. Если белок CheW характеризовался весьма высокой инвариантностью первичной аминокислотной последовательности, то в случае PspA требовался программный расчёт консенсусного сиквенса. Созданию синтетических генов также предшествовала необходимость учёта современных принципов оптимизации триплетов для обеспечения их эффективной экспрессии в клетках E. coli. В рамках этого осуществляли оптимизацию кодонового состава генов, кодирующих нативный CheW и консенсусный PspA. В результате разработанные in silico оптимизированные варианты гибридных генов стали основой для дальнейшей практической работы (рис. 1).
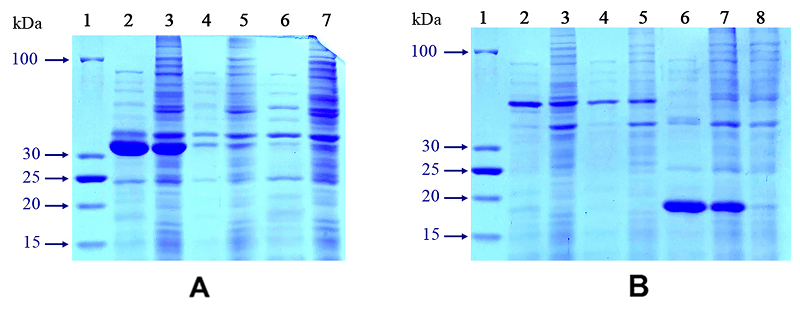
Конструирование экспрессионных векторов и молекулярное клонирование Для сборки спланированных конструкций использовали метод Гибсона, основанный на ДНК рекомбинации, позволяющей поэтапно собрать несколько перекрывающихся фрагментов ДНК в единую молекулу. В работе плазмидный вектор рЕТ-21а («Novagen», США) предварительно линеаризовали, а затем использовали для рекомбинации с заранее подготовленными синтетическими олигонуклеотидами. Для этого к полученной эквимолярной смеси добавляли 10 мкл 2-кратного мастер-микса (см. раздел Методика). Реакционную смесь инкубировали при 50оС. На следующем этапе проводили трансформацию компетентных клеток E. coli 10 мкл полученной лигазной смеси. Правильность сборки генно-инженерных конструкций на разных этапах подтверждали рестрикционным картированием, ПЦР с соответствующими праймерами (Cw-F: catatgaaaaccctggcggatgcgctgaaaga; Cw-R: acctagctcattcagggctgcgtctaactctttct; Pas-F: atgaaagaaggcctggaacaggcgatta; Pas-R: gccagtttcacgccttctttcacggta) и секвенированием. Таким образом, были созданы рекомбинантные плазмиды pET-PspA-sp-CheW и pET-CheW-sp-PspA, в которых гены интереса находятся под контролем высокоэффективных регуляторных элементов бактериофага Т7 (рис. 1). Изучение экспрессии PspA-sp-CheW и CheW-sp-PspA Компетентные клетки лабораторного штамма E. coli BL21(DE3) pLysS трансформировали экспрессионными плазмидами, созданными на предыдущем этапе, а также плазмидой pET-TpeCheW, кодирующей исходный термостабильный белок CheW в качестве одного из контролей. Анализ результатов экспрессии гибридных генов в клетках E. coli при помощи электрофореза в 12% ДСН-ПААГ продемонстрировал появление белковой полосы ожидаемого молекулярного веса 34 кДа в лизатах индуцированных клеток, содержащих плазмиду pET-CheW-sp-PspA (рис. 2А). В случае же белка PspA-sp-CheW наблюдается лишь слабо выраженная полоса на уровне, соответствующем примерно 70 кДа, что может свидетельствовать о формировании артефактной димерной формы (рис. 2В).
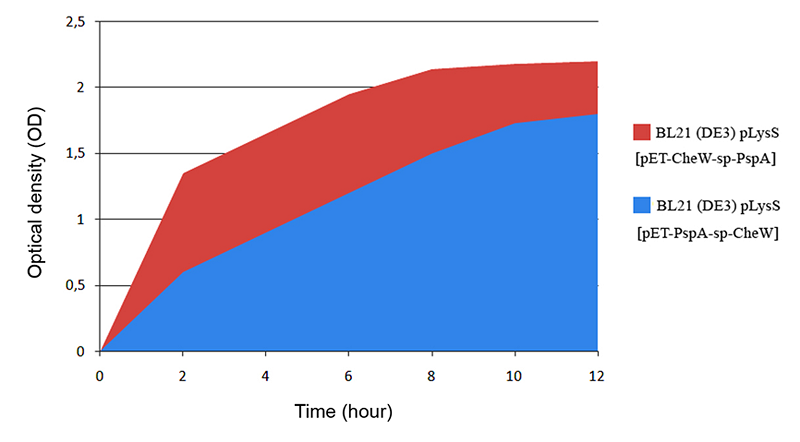
Денситометрический анализ электрофореграмм показал, что уровень экспрессии белка CheW-sp-PspA составил не менее 27% от общего белка клетки, в то время как экспрессия PspA-sp-CheW не превышала 6%. Выход PspA-sp-CheW и CheW-sp-PspA из 16-18 часовых культур клеток E. coli составил около 40 мг и 230 мг с одного литра культуральной среды соответственно. Необходимо также отметить, что рост клеток с плазмидой pET-PspA-sp-CheW до и после индукции существенно отличался от такового для клеток, трансформированных плазмидой pET-CheW-sp-PspA, что наглядно демонстрирует график, отражающий динамику накопления биомассы продуцентов (рис. 3). Соотнося эти данные с итоговым уровнем экспрессии PspA-sp-CheW, можно сделать вывод, что данный белок плохо переносится клетками E. coli, обладая, по всей видимости, определённой токсичностью для данного микроорганизма.
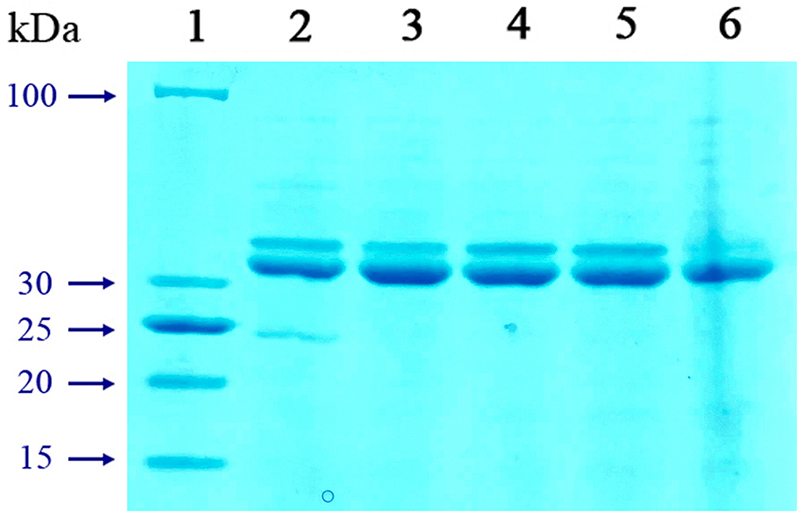
Определение оптимального диапазона pH и температуры Важнейшим показателем для сконструированных белков является их термостабильность, поэтому основной фазой данного этапа исследования стал сбор супернатанта, который должен был включать наиболее термоустойчивые и растворимые белковые фракции. При этом только химерный белок CheW-sp-PspA обладал высоким уровнем термостабильности и локализовался в основном в растворимой фракции клеточного лизата (рис. 2, 4). Попутно было определено влияние pH на термостойкость при различных температурах. Картина электрофореза CheW-sp-PspA была идентичной для всех диапазонов pH от 5.7 до 8.5 (показаны при pH 7.5). рН не оказывал существенного влияния на термостабильность, что даёт возможность в дальнейшем использовать этот белок с различными буферными системами и физиологическими значениями рН.
Простое выделение и очистка белка Для очистки белков осадки индуцированных клеток E. coli суспендировали в специальном буфере для термолизиса из расчёта 10% от объёма культуры. Клеточную суспензию обрабатывали ультразвуком в интенсивном режиме на льду (см. раздел Методика). Образцы нагревали при температуре ≥80°C в течение 40 мин, после чего клеточный дебрис удаляли центрифугированием. К оставшемуся супернатанту добавляли кристаллический (NH4)2SO4 до насыщения 80% и оставляли смесь на 20 ч при 4оС для концентрирования рекомбинантных белков. Раствор осветляли центрифугированием при 6800 g не менее 5 мин, осадок либо лиофилизировали, либо растворяли в буфере (20 мМ Трис-HCl; рН 7.5; 10 мМ ЭДТА; 10% глицерина) и хранили при температуре -20°C до дальнейшего использования. ЗАКЛЮЧЕНИЕ Спланированы и сконструированы индуцибельные синтетические генно-инженерные конструкции, позволяющие получать в гетерологических системах химерные белки, объединяющие в различной пространственной ориентации копию адаптивного полипептида CheW T. petrophila и копию иммуногенного B-региона поверхностного антигена из S. pneumoniae. Введение линкеров в зоны объединения CheW и PspA призвано обеспечить необходимую гибкость итоговой рекомбинантной конструкции и правильный фолдинг гибридного белка, позволяющие получить его в растворимой форме. Экспериментально было продемонстрировано, что пептид CheW способен выступать в роли весьма эффективной системы слияния. При этом полученный С-концевой гомолог гибридного белка обладал рассчитанным молекулярным весом 34 кДа, высоким уровнем экспрессии (не менее 27% от общего белка) и необходимым уровнем термостабильности, обеспечивающим отсутствие денатурации белка в течение 40-минутной экспозиции в пределах от 75 оС до 99оС. Полученный образец имел степень чистоты, превышающую 90%. Таким образом, созданный химерный белок CheW-sp-PspA обладает всеми необходимыми и достаточными свойствами, чтобы в дальнейшем стать основой для разработки новых кандидатных диагностикумов и профильных рекомбинантных вакцин. СОБЛЮДЕНИЕ ЭТИЧЕСКИХ СТАНДАРТОВ Работа не содержит каких-либо исследований с участием людей или использованием животных в качестве объектов. БЛАГОДАРНОСТИ На некоторых этапах исследования было использовано оборудование Центра коллективного пользования ФГБНУ НИИВС им. И.И. Мечникова (№ 075-15-2021-676). ФИНАНСИРОВАНИЕ Работа выполнена в рамках Программы фундаментальных научных исследований в Российской Федерации на долгосрочный период (2021-2030 годы) (№122022800499-5). КОНФЛИКТ ИНТЕРЕСОВ Авторы заявляют об отсутствии конфликта интересов. ЛИТЕРАТУРА
|