|
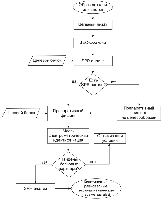
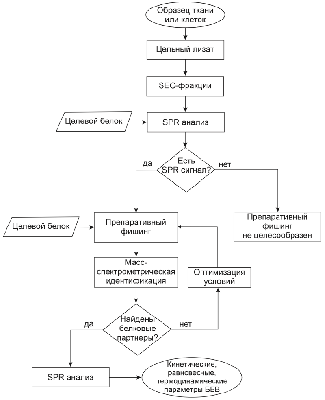
СОДЕРЖАНИЕ 1. СТРУКТУРА ЭКСПЕРИМЕНТАЛЬНОЙ ПЛАТФОРМЫ 2. ПРИМЕРЫ ИСПОЛЬЗОВАНИЯ ЭКСПЕРИМЕНТАЛЬНОЙ ПЛАТФОРМЫ ДЛЯ ИССЛЕДОВАНИЯ СУБИНТЕРАКТОМОВ ЦЕЛЕВЫХ БЕЛКОВ СОБЛЮДЕНИЕ ЭТИЧЕСКИХ СТАНДАРТОВ Рисунок 2Блок-схема экспериментальной платформы для исследования белковых субинтерактомов. Таблица 1Структура экспериментальной платформы для исследования субинтерактомов целевых белков. |
Стратегия экспериментальных исследований интерактомики целевых белков
Научно-исследовательский инстиут биомедицинской химии имени В. Н. Ореховича, Ключевые слова: белок-белковые взаимодействия; аффинное выделение; белковый субинтерактом; комплексообразование; поверхностный плазмонный резонанс; платформа DOI: 10.18097/BMCRM00224 ВВЕДЕНИЕ Межмолекулярные взаимодействия между белками и пептидами, а также их взаимодействия с эндогенными и экзогенными метаболитами, играют ключевую роль в осуществлении и регуляции биологических процессов и передачи внутриклеточных сигналов [1, 2]. Информация о межмолекулярных взаимодействиях расширяет понимание структурно-функциональных связей на всех уровнях организации живых систем. В настоящее время не вызывает сомнения тот факт, что большинство белков функционируют в составе различных молекулярных комплексов, формирование которых происходит за счёт белок-белковых взаимодействий (ББВ). Их совокупность применительно к биологическому контексту можно определить как «белковый интерактом». Соответственно под термином «субинтерактом» понимается совокупность ББВ какой-либо биологической ткани или органа, клеточной линии или взаимодействия конкретного целевого белка с его белковыми партнёрами, взаимодействующими с ним напрямую или опосредованно [3]. Исследования белковых субинтерактомов очень важны для изучения функций и механизмов регуляции неизвестных или плохо аннотированных белков, понимания архитектуры внутриклеточных молекулярных машин, а также дизайна модуляторов ББВ [4]. Ранее мы применяли комбинации экспериментальных подходов, а также аналитических и препаративных методов для изучения субинтерактомов разных в функциональном отношении клеточных белков, что позволило идентифицировать белковые субинтерактомы простациклин синтазы (PTGIS) [3], тромбоксансинтазы (TBXAS1) [5], феррохелатазы [6] и транскрипционного фактора CXXC1, содержащего ДНК-связывающий домен «цинковые пальцы» [7]. Целью данной работы была концептуализация принципов разработанной нами экспериментальной платформы для исследования белковых субинтерактомов и подробное описание её особенностей. 1. СТРУКТУРА ЭКСПЕРИМЕНТАЛЬНОЙ ПЛАТФОРМЫ Экспериментальная платформа для исследования белковых субинтерактомов основана на использовании комбинации четырёх методов: 1) поверхностный плазмонный резонанс (SPR, от англ. Surface Plasmon Resonance); 2) аффинная хроматография; 3) высокоэффективная эксклюзионная хроматография (SEC, от англ. Size Exclusion Chromatography); 4) жидкостная хроматография, совмещенная с тандемной масс-спектрометрией (LC-MS/MS, от англ. Liquid Chromatography - Tandem Mass Spectrometry). Экспериментальная платформа состоит из пяти основных блоков (таблица 1): I. Получение тканевых или клеточных лизатов, II. Аналитический молекулярный фишинг, III. Препаративный молекулярный фишинг, IV. Фракционирование белкового материала лизата по молекулярной массе, V. Верификация межмолекулярных взаимодействий. Общая схема экспериментальной платформы приведена на рисунке 1.
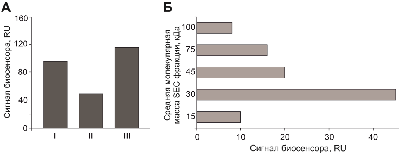
1.1. Получение тканевых или клеточных лизатов (блок I) На сегодняшний день известно большое число протоколов получения лизатов из разных биологических материалов [8, 9], однако универсальной методики получения лизата для выполнения интерактомного анализа нет. Отсутствие стандартизации данной процедуры затрудняет сравнительный анализ интерактомных данных из разных источников. Следует отметить, что выбор метода получения лизата в значительной степени определяет представленность индивидуальных белков и белковых комплексов в полученных образцах. Выбор оптимального состава лизирующего буфера и методики получения лизата обусловлен поиском компромисса между такими факторами, как эффективность экстракции белков из биологического материала, предотвращение денатурации и агрегации белкового материала, сохранение нативной структуры индивидуальных белков и стабильных белковых комплексов, а также подавление фоновой протеолитической активности. Лизирующие буферы, содержащие физиологические концентрации солей с добавлением «мягких» неионных детергентов (типа Тритон X-100 или CHAPS), пригодны для экстракции растворимых цитоплазматических и частично мембраносвязанных белков. Более «жёсткие» условия экстракции белков в присутствие высоких концентраций солей и сильных детергентов направлены на получение лизатов с более высоким содержанием белков, в том числе мембранных и белков субклеточных частиц [10]. Однако при таком подходе происходит резкое снижение доли нативных белковых комплексов, сохранение которых критично для интерактомных исследований. Обратимое изменение рН лизата в кислую сторону примерно до рН 4,0 с последующей нейтрализацией может быть использовано для повышения эффективности аффинного выделения индивидуальных белковых партнёров целевого белка. Так, в наших исследованиях кратковременное закисление с последующей нейтрализацией тканевого лизата приводило к диссоциации стабильных белковых комплексов; при этом не было значимого снижения уровней комплексообразования модельных белковых пар [3, 11]. С помощью SPR анализа было показано, что связывание белкового материала лизата с иммобилизованным на оптическом чипе целевым белком было значительно выше в «раскисленном» лизате по сравнению с интактным. Это указывает на появление в лизате дополнительного количества свободных молекул белковых партнёров, находившихся ранее в составе стабильных белковых комплексов и недоступных для взаимодействия с целевым белком [3, 11]. 1.2. Аналитический молекулярный фишинг (блок II) Аналитический молекулярный фишинг (от англ. fishing, рыбалка) позволяет выполнить моделирование и оптимизацию протокола [12] препаративного молекулярного фишинга (блок III). Для этого целевой белок иммобилизуют (ковалентно или нековалентно) на поверхности оптического чипа SPR биосенсора в качестве лиганда. Далее через измерительную ячейку биосенора пропускают образец лизата и регистрируют уровень связывания белкового материала с иммобилизованным целевым белком. После окончания инжекции лизата регистрируют кинетику диссоциации комплексов, по которой можно рассчитать константу скорости диссоциации (koff) и период полураспада (τ = ln2/koff). Стабильные белковые комплексы характеризуются медленной кинетикой диссоциации (koff ≤ 0,01 c-1), тогда как лабильные белковые комплексы диссоциируют гораздо быстрее. Более подробная интерпретация результатов аналитического молекулярного фишинга как модели препаративного фишинга, в частности, возможностей интерпретации сенсограмм взаимодействия белкового материала лизата с целевым белком, представлена в нашем тематическом обзоре [12]. В случае отсутствия сигнала связывания белкового материала лизата с иммобилизованным на чипе целевым белком делается заключение о нецелесообразности дальнейшего выполнения процедуры препаративного фишинга в данных условиях. В лизатах биологического материала наряду с индивидуальными белками и стабильными белковыми комплексами часто присутствуют протеолитические фрагменты белков, которые могут взаимодействовать с целевым белком и обуславливать ложноположительные результаты SPR анализа. Это подтверждается масс-спектрометрическим анализом SEC-фракций лизата со средней молекулярной массой 10-15 кДа, в которых обнаруживаются фрагменты высокомолекулярных белков [13]. Поэтому для исключения ложноположительных результатов SPR анализа мы выполняли предварительное отделение низкомолекулярной SEC-фракции лизата (<15 кДа) на центрифужных пробирках с фильтром типа Viva spin (“Sartorius AG”, Германия). Дополнительно для блокирования протеолитической активности при получении лизата и в ходе препаративного молекулярного фишинга в лизирующий буфер добавляется «коктейль» ингибиторов протеолитических ферментов. 1.3. Препаративный молекулярный фишинг (блок III) Препаративный молекулярный фишинг представляет собой комбинацию двух методов: 1) хроматографическое выделение из лизата белковых партнёров на аффинной колонке с иммобилизованным целевым белком в качестве лиганда; 2) масс-спектрометрическая идентификация белков, элюированных с аффинной колонки [14-16]. В отличие от разнообразных сложных методов молекулярного фишинга, основанных на использовании различных меток и генно-инженерных конструкций, данный вариант молекулярного фишинга основан на выделении белковых партнёров из лизата за счёт их прямого связывания с иммобилизованным целевым белком. Для исключения ложноположительных результатов за счёт неспецифической сорбции высоко адгезивных белков выполняется контрольный эксперимент в виде аналогичной хроматографической процедуры с использованием сорбента без иммобилизованного целевого белка. Данный вариант препаративного молекулярного фишинга позволяет выделять белковые партнёры, которые формируют с целевым белком стабильные долгоживущие комплексы с длинным периодом полураспада. Нужно также отметить, что при выполнении данной процедуры на аффинной колонке с иммобилизованным целевым белком одновременно «ловятся» не только прямые белковые партнёры, но и различные мультимерные белковые комплексы и молекулярные агрегаты, в которых содержащийся прямой партнёр выполняет роль специфической аффинной метки. Подобные комплексы могут содержать как партнёры второго и далее уровней (партнёры партнёров), так и абсолютно посторонние белки, не имеющие отношения к интерактомике целевого белка. Данный побочный эффект молекулярного фишинга является источником ложноположительных результатов при экспериментальном выделении прямых партнёров целевого белка. Для решения этой проблемы был разработан подход предварительной очистки лизата от крупных комплексов и агрегатов путём последовательного выполнения следующих процедур: 1) разделение лизата на множество фракций с разными молекулярными массами путём его фракционирования с помощью высокоэффективной SEC хроматографии; 2) анализ каждой фракции лизата на присутствие потенциальных партнёров целевого белка с помощью аналитического молекулярного фишинга (последовательный SPR анализ взаимодействия белкового материала фракций с иммобилизованным на оптическом чипе целевым белком); 3) получение очищенного от крупных молекулярных комплексов и агрегатов лизата путём объединения тех фракций, в которых аналитический фишинг показал максимальные сигналы связывания с целевым белком, и исключением фракций с молекулярными массами более 100 кДа; 4) выполнение препаративного молекулярного фишинга потенциальных партнёров на аффинной колонке с иммобилизованным целевым белком из очищенного и обогащённого лизата. При использовании метода прямого аффинного выделения потенциальных партнёров целевых белков возможно появление и ложноотрицательных результатов, связанных с тем, что прямые белковые партнёры могут находиться в составе стабильных белковых комплексов и быть физически недоступны для взаимодействия с иммобилизованным на сорбенте целевым белком. Данная проблема решается путём диссоциации стабильных белковых комплексов при кратковременном закислении лизата с последующей нейтрализацией избытка кислоты. Данный подход не нов, рН-зависимая диссоциация молекулярных комплексов для высвобождения связанных белковых партнёров применяется, например, при регенерации биосенсорных чипов [17] и аффинных сорбентов, а также при анализе иммуногенности лекарственных препаратов [18, 19]. Следующим важным этапом является верификация ББВ между целевым белком и идентифицированными потенциальными белковыми партнёрами. Как правило, с помощью прямого препаративного фишинга в лизате биологического материала можно найти от единиц до нескольких десятков потенциальных белковых партнёров целевого белка. Однако верификация каждого потенциального белкового партнёра встречает большие сложности в силу ограниченности источников получения и дороговизны высокоочищенных рекомбинантных белковых препаратов. Помимо описанного выше прямого препаративного молекулярного фишинга белковых партнёров на поверхности аффинного сорбента с иммобилизованным целевым белком, возможен и непрямой препаративный фишинг, когда целевой белок добавляется в лизат и после инкубации выделяется вместе со связавшимися с ним партнёрами [20]. Для этого к лизату добавляется целевой белок с аффинной меткой (например, биотинилированный целевой белок) и инкубируется для специфического связывания с партнёрами. Далее выполняется аффинное выделение целевого белка в комплексах с партнёрами на аффинном сорбенте (в случае биотинилированного белка – на сорбенте с иммобилизованным стрептавидином). Данный вариант направлен на снижение числа неспецифически выделяемых из лизата белков и может быть дополнен процедурой рН-зависимой диссоциации стабильных белковых комплексов. С технической точки зрения, аффинный сорбент получают путём ковалентной иммобилизации целевого белка за свободные аминогруппы остатков лизина на поверхности инертного сорбента на основе CNBr-сефарозы-4B. Далее аффинный сорбент помещают в колонку хроматографа для последующего пропускания цельного лизата или определённых SEC-фракций лизата. Связавшиеся с аффинным сорбентом белки лизата, далее элюируют с колонки, проводят трипсинолиз и масс-спектрометрическую идентификацию. 1.4. SEC хроматография белкового материала лизата (блок IV) Сочетание SEC хроматографии белкового материала лизата и LC-MS/MS анализа применяют для профилирования состава стабильных белковых комплексов [21-23], вплоть до выделения отдельных мультимерных комплексов [24, 25]. Это позволяет выявить кофракционированные белки и вероятные партнёры по ББВ согласно совпадению их профилей количественного или полуколичественного распределения в SEC-фракциях [26]. Сочетание SEC хроматографии лизата и SPR анализа фракций лизата позволяет решать методическую задачу по полуколичественной оценке белкового материала лизата, который мог бы специфично связываться с целевым белком. Так, в качестве примера на рисунке 2 показаны результаты SPR анализа; приведены сигналы связывания белкового материала разных SEC-фракций лизата печени человека с иммобилизованным на оптическом чипе биосенсора транскрипционным фактором CXXC1 (белок, кодируемый геном, локализованным в 18 хромосоме человека). На рисунке 2 видно, что SEC-фракции лизата со средними молекулярными массами от 30 кДа до 75 кДа являются основными “донорами” белков, взаимодействующими с CXXC1. Таким образом, для увеличения доли специфичных взаимодействий целевого белка с его прямыми партнёрами при последующем выполнении препаративного молекулярного фишинга предпочтительным будет выбор тех SEC-фракций лизата, которые соответствуют интервалу максимального связывания белкового материала лизата с целевым белком.
SEC хроматография лизатов может также быть применена для изучения модулирующего действия биологически активных веществ (БАВ) на ББВ в стабильной части белкового интерактома. Сравнительный количественный анализ белков в SEC-фракциях контрольного образца лизата и опытного образца, к которому был добавлен БАВ, позволяет выявить изменения в составе стабильных белковых комплексов, произошедшие в присутствии БАВ. В результате могут быть идентифицированы целевые белки и белковые комплексы, являющиеся возможными мишенями действия БАВ [27]. Однако, для успешной реализации данного подхода требуется выполнение большой предварительной работы по оптимизации ряда параметров, таких как растворимость БАВ в лизирующем буфере, его конечная концентрация в лизате, условия и время инкубации. 1.5. SPR верификация потенциальных ББВ (блок V) SPR анализ позволяет верифицировать взаимодействие с целевым белком потенциальных белковых партнёров, идентифицированных с помощью препаративного молекулярного фишинга. В результате SPR верификации потенциальные белковые партнёры могут быть разделены на две группы: 1) прямые белковые партнёры, формирующие с целевым белком стабильные комплексы; 2) потенциальные белковые партнёры второго порядка (партнёры партнёров), которые не взаимодействуют напрямую с целевым белком, но вероятно могут взаимодействовать с прямыми партнёрами, так как они были выделены из лизата в составе мультимерных комплексов, содержащих белки первой группы. На втором этапе выполняется подробный SPR анализ взаимодействия выявленных прямых белковых партнёров с целевым белком с количественной оценкой кинетических параметров ББВ (константы скоростей ассоциации и диссоциации комплексов), аффинности (равновесная константа диссоциации комплексов) и даже термодинамики взаимодействия [28]. Стоит заметить, что наряду с SPR анализом для верификации возможных ББВ могут быть использованы альтернативные методы для анализа, такие как иммунопреципитация [29, 30], термофорез [31] и другие [32]. 2. ПРИМЕРЫ ИСПОЛЬЗОВАНИЯ ЭКСПЕРИМЕНТАЛЬНОЙ ПЛАТФОРМЫ ДЛЯ ИССЛЕДОВАНИЯ СУБИНТЕРАКТОМОВ ЦЕЛЕВЫХ БЕЛКОВ 2.1. Исследование субинтерактома феррохелатазы Митохондриальная феррохелатаза (FECH), катализирует процесс внедрения иона железа в протопорфирин IX для формирования гема, являющегося кофактором многих белковых молекул. Данный белок кодирует ген FECH, расположенный в локусе 18 хромосомы человека. Выбор FECH в качестве целевого белка для интерактомных исследований был обусловлен работой в рамках международной программы «Протеом человека» (Российская Федерация участвует в данном научном проекте и объектами её исследований являются белки, кодируемые генами 18 хромосомы). Исследование FECH представляет также определенный интерес для медицины. Имеются данные о роли FECH в возрастной неоваскуляризации в тканях глаза [33] и о возможности воздействия на FECH малыми молекулами [34]. Фермент FECH является гомодимером [35] и его субинтерактом может включать в себя более 50 потенциальных белковых партнёров, участвующих в процессах метаболизма гема, организации митохондрий, генной экспрессии и производстве энергии [36]. Таким образом, выяснение всего спектра белковых партнёров FECH поможет более детально понять метаболизм гема и сопряженные с ним процессы. Целью исследования было выявление новых белковых партнёров FECH методом препаративного молекулярного фишинга. Для этого готовили образцы цельного лизата ткани печени человека с использованием лизирующего буфера CelLytic™ MT Cell Lysis Reagent [37]. Далее выполняли SEC хроматографию лизата с отбором фракций, содержащих белковый материал с молекулярной массой от 15 кДа до 100 кДа. Эти фракции объединяли и использовали для препаративного молекулярного фишинга. Феррохелатаза была ковалентно иммобилизована на инертном сорбенте CNBr-сефарозе-4B (“GE Healthcare”, США) за аминогруппы белка согласно протоколу производителя. Аффинный сорбент помещали в специальную колонку хроматографа и пропускали образец объединённых SEC-фракций лизата. Для коррекции результатов фишинга на возможную неспецифическую сорбцию белков на сорбенте выполняли контрольный эксперимент с пропусканием того же образца лизата через колонку с сорбентом без иммобилизованной FECH. Связавшиеся на колонках белки элюировали, выполняли их трипсинолиз и масс-спектрометрическую идентификацию [37]. Среди белковых партнёров была выявлена митохондриальная NADPH-зависимая адренодоксин редуктаза (ADR), которая участвует в электронтранспортных процессах, и кодируется геном FDXR, локализованным на 17 хромосоме человека. ADR катализирует восстановление NADP, используя в качестве донора электронов малый белок ферредоксин, содержащий железосерный кластер [38]. SPR анализ взаимодействия ADR (в качестве аналита) с иммобилизованным на оптическом чипе FECH (в качестве лиганда) показал, что в условиях in vitro эти два белка образуют медленно-ассоциируемый комплекс (kon <1000 M-1c-1) с равновесной константой диссоциации (Kd) порядка 10-5 М [6]. При этом взаимодействие ADR происходит преимущественно с мономерной формой FECH [39]. Таким образом, данная работа продемонстрировала пример эффективной реализации экспериментальной интерактомной платформы по нахождению в лизате биологического материала неизвестного ранее прямого белкового партнёра, взаимодействующего с целевым белком (FECH), что было подтверждено SPR верификацией. 2.2 Исследование субинтерактомов тромбоксансинтазы и простациклинсинтазы Тромбоксансинтаза (TBXAS1) и простациклинсинтаза (PTGIS) являются мембраносвязанными ферментами, участвующими в метаболизме простаноидов [40], которые связываются со своими рецепторами на поверхности клеток и регулируют как физиологические процессы в организме человека, так и принимают участие в формировании патологических состояний [41]. Целью исследования был сравнительный анализ белковых субинтерактомов PTGIS и TBXAS1, которые являются функциональными антагонистами [5]. Для этого были приготовлены лизаты из шести разных тканей крысы (аорта, лёгкое, мозг, печень, сердце и семенники) с использованием лизирующего буфера CelLytic™ MT Cell Lysis Reagent [5]. Каждый образец лизата был протестирован описанным выше методом аналитического фишинга на SPR биосенсоре на предмет связывания белкового материала с иммобилизованными на оптическом чипе PTGIS и TBXAS1. Положительным результатом аналитического фишинга считали сигналы биосенсора, превышающие 10 RU. Для выполнения прямого препаративного фишинга потенциальных белковых партнёров были приготовлены два аффинных сорбента с иммобилизованными PTGIS и TBXAS1 на CNBr-сефарозе-4B. В качестве контроля для коррекции результатов на возможную неспецифическую сорбцию белков лизата был использован пустой сорбент без иммобилизованных белков. В результате, из всех шести цельных лизатов были выделены 36 потенциальных белковых партнёров: 11 и 25 для TBXAS1 и PTGIS, соответственно. Среди них были семь общих белковых партнёров (CALR, CANX, HSP90B1, PDIA6, GRPEL1, HSPA1L, HSPA2), функционирующих в качестве шаперонов. Кроме того, были обнаружены 3 и 10 белковых партнёров TBXAS1 и PTGIS, соответственно, которые были выделены только из лизата одной ткани, что указывает на существование тканеспецифичных ББВ [5]. Для верификации бинарного ББВ был выбран цитохром P450 2E1 (CYP2E1), который был идентифицирован как потенциальный белковый партнёр обоих целевых белков. Для этого PTGIS и TBXAS1 были ковалентно иммобилизованы на оптическом чипе в качестве лигандов, а растворы CYP2E1 в разных концентрациях инжектировали через измерительные ячейки биосенсора в качестве аналитов. Результаты SPR анализа показали, что CYP2E1 взаимодействует как с TBXAS1, так и с PTGIS. Рассчитанные из набора сенсограмм значения Kd комплексов CYP2E1 с TBXAS1 и PTGIS были порядка 10-7 М [3, 42]. Таким образом, данная работа также подтвердила эффективность реализации экспериментальной интерактомной платформы по нахождению в лизате биологического материала неизвестного ранее прямого белкового партнёра, что было подтверждено последующей SPR верификацией. ЗАКЛЮЧЕНИЕ Экспериментальная платформа для исследований субинтерактомов целевых белков основана на применении комбинации четырёх методов: 1) поверхностного плазмонного резонанса; 2) аффинной хроматографии; 3) высокоэффективной эксклюзионной хроматографии и 4) белковой масс-спектрометрии. Платформа позволяет получать первичную информацию в виде перечня потенциальных белковых партнёров, формирующих стабильные комплексы с целевыми белками. С помощью SPR анализа осуществляется верификация потенциальных партнёров путём анализа их прямого взаимодействия с целевыми белками и количественной оценкой кинетических и равновесных параметров этих ББВ. Идентификация неизвестных ранее белковых партнёров целевых белков имеет важное значение в расширении знаний о функциях как целевых белков, так и предсказании способов регуляции ББВ с их участием как возможных мишеней для создания лекарств нового поколения. СОБЛЮДЕНИЕ ЭТИЧЕСКИХ СТАНДАРТОВ Настоящая статья не содержит каких-либо исследований с участием людей или с использованием животных в качестве объектов. БЛАГОДАРНОСТИ Высокоочищенные рекомбинантные препараты CXXC1, FECH, PTGIS и TBXAS1 были наработаны и любезно предоставлены Институтом биоорганической химии НАН Беларуси. ФИНАНСИРОВАНИЕ Работа выполнена в рамках Программы фундаментальных научных исследований в Российской Федерации на долгосрочный период (2021-2030 годы) (№122030100168-2). КОНФЛИКТ ИНТЕРЕСОВ Авторы заявляют об отсутствии конфликта интересов. ЛИТЕРАТУРА
|